(2: 常州工程职业技术学院制药与环境工程学院, 常州 213161)
(3: 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008)
(4: 江苏省地理信息资源开发与利用协同创新中心, 南京 210023)
(2: Institute of Pharmaceutical and Environmental Engineering, Changzhou Vocational Institute of Engineering, Changzhou 213164, P. R. China)
(3: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
(4: Jiangsu Center for Collaborative Innovation in Geographical Information Resource Development and Application, Nanjing 210023, P. R. China)
太湖是中国五大淡水湖之一.生活污水排放与过度施用氮肥的农田径流导致太湖水体营养物质浓度升高而形成富营养化, 大量的有机质、氮、磷等营养物质在湖泊沉积物中沉降富集[1-3].太湖污染物从西北部进入, 经过水体稀释, 流向东南方向, 最终主要从与东太湖相连的太浦河出太湖[4].因此, 太湖从西北向东南, 湖区富营养化程度逐渐下降, 不同湖区的水质状况不完全相同, 主要的环境因子也呈现一定浓度梯度[5].
太湖中部分外源无机氮以及由有机氮矿化的无机氮可以通过硝化与反硝化偶联过程转化成N2被去除[6].其中, 硝化作用是氮循环的核心环节, 硝化过程由硝化微生物执行.硝化微生物包括氨氧化细菌(AOB)、氨氧化古菌(AOA)以及亚硝酸盐氧化细菌(NOB), 它们不仅是氨氮转化的主要驱动者, 也是反硝化作用底物硝态氮(NO3--N)的生产者.氨氧化过程被认为是硝化过程的限速步骤.目前, 湖泊硝化微生物的研究多以沉积物为研究对象, 且研究多集中于AOA与AOB的分布、群落组成及环境影响因素研究[6-8], 而环境中的NOB由于缺乏分析手段, NOB的研究经常被忽视.随着高通量测序技术的出现, NOB越来越受关注[9], 有研究发现硝化螺菌属(Nitrospira)能够将NH3直接氧化成NO3--N[10].影响湖泊水体和底泥AOA和AOB丰度的环境因子差异较大, 水体中氨氧化微生物丰度通常受铵态氮(NH4+-N)、DO和pH值的影响[11];底泥的AOA丰度主要影响因素有pH、NH4+-N、NO3--N、总氮(TN)、总磷(TP)和总溶解性碳[12-13], AOB丰度主要受NH4+-N、pH和NO3--N的影响[8].我国淡水湖泊沉积物中AOA丰度高于AOB丰度[6, 8, 14], 但在高原淡水湖中沉积物AOB丰度高于AOA丰度[15], 其原因不完全清楚.氨氧化产生的亚硝态氮(NO2--N)经NOB的作用被迅速氧化成NO3--N, 湖泊沉积物中NOB丰度和分布主要受NH4+-N和NO2--N的影响[16-18].目前, 关于不同富营养化水平的淡水湖泊水体和沉积物中氨氧化微生物、亚硝酸盐氧化微生物的分布、群落组成和丰度等多角度的研究报道比较少见.
太湖北部梅梁湾湖区和湖心区水体富营养化程度不同.梅梁湾是太湖污水的主要入流区之一, 是太湖典型的富营养化湖区, 该湖区具有藻型湖区特征, 水体悬浮物浓度高于湖心区, 并频繁暴发大面积的蓝藻水华[4, 8];而湖心区水体则轻度富营养化.太湖梅梁湾与湖心区水体与沉积物中环境条件不同, 硝化微生物分布、群落组成和丰度也可能不同.本研究采集梅梁湾和湖心区内不同深度水体样品和沉积物样品, 分析水体和沉积物样品中AOA、AOB和NOB的分布和群落组成和丰度, 研究太湖不同富营养化水平水体和沉积物中硝化微生物分布和群落组成特征, 并探寻驱动硝化微生物分布与群落丰度的关键环境因子.本研究有助于更全面了解太湖生态系统氮素循环及相关功能微生物, 为太湖环境保护和生态修复提供理论依据.
1 材料与方法 1.1 样品采集于2015年11月5日分别在太湖北部梅梁湾和湖心区各选取1个位点分别标记为HT(31°27′20″N, 120°11′23″E)、ET(31°17′22″N, 120°10′56″E)(图 1).水样采集使用有机玻璃采水器, 分别从表层、中层和底层采集3个重复水样, 具体采样位置为:梅梁湾湖区水面下0.53、1.03和2.11 m处水样(分别记为HT-U、HT-M和HT-B);湖心区水面下0.52、1.07和2.18 m处水样(分别记为ET-U、ET-M和ET-B).用柱状采泥器采集30 cm深度柱状沉积物, 在2个采样区域各采集3个不同位点的沉积物样品, 沉积物现场分割, 取0~2 cm表层样品保存于灭菌袋中, 并冲入氮气后迅速封口, 作为沉积物样品(分别命名为HT-S和ET-S).水样和沉积物样品采集后于冷藏条件下运回实验室进行处理和保藏.水样采集后24 h内通过0.22 μm微孔滤膜(Whatman GmbH, Germany)过滤, 滤液用于水体理化指标测定, 滤膜残渣保存在-80℃冰箱中用于分子生物学分析[19].每个沉积物样品分成两份, 一份用于理化指标测定, 一份保存在-80℃用于分子生物学分析.
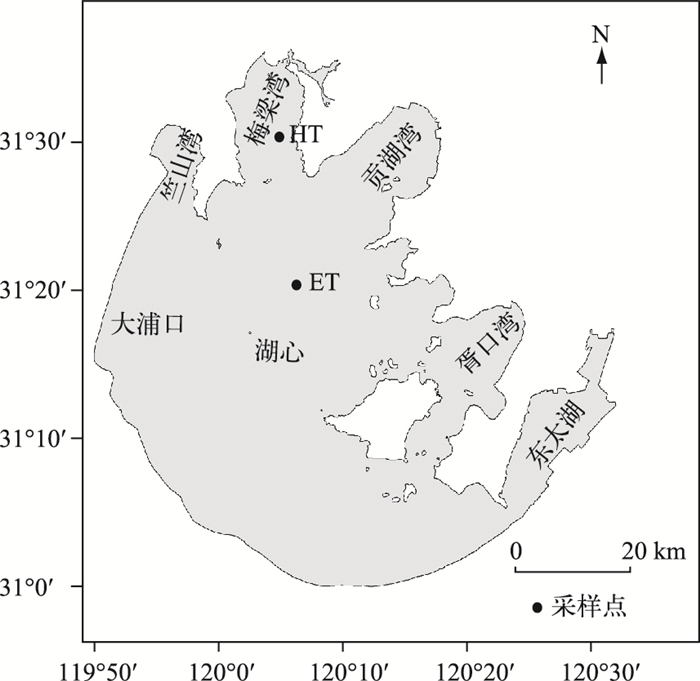
|
图 1 太湖水体和沉积物样品采集位置示意 Fig.1 Sampling sites of water and sediment in Lake Taihu |
不同深度水体的温度、DO和pH值等理化指标使用多参数水质仪(YSI 6600 V2, USA)现场测定;水体中的无机氮(包括NH4+-N、NO3--N和NO2--N)浓度用流动分析仪(Skalar San Plus, Netherlands)测定;水体叶绿素a(Chl.a)、TP、TN浓度、高锰酸盐指数(CODMn)和透明度(SD)的测定方法参见文献[20].
沉积物中的无机氮采用2 mol/L KCl按5 :1(v/m)的水土比浸提, 在20℃、200转/min条件下振荡1 h后过滤, 滤液无机氮含量的测定方法同水样;沉积物用蒸馏水按2.5 :1(v/m)的水土比浸提, 振荡10 min并静置30 min, 采用台式pH计(Mettler-Toledo)测定悬浊液pH值;沉积物DO值测定采用溶氧微电极(Unisense Company, Denmark)测定;沉积物TP、TN与总有机碳(TOC)的测定方法参见文献[12, 21].
1.3 水体和沉积物总DNA提取沉积物与滤膜残渣总DNA采用Fast DNA © SPIN Kit for soil(MP Biomedicals, USA)试剂盒提取, 具体操作方法参照说明书, 提取后的总DNA溶解于100 μl TE缓冲液中. DNA浓度和纯度采用NanoDrop 2000分光光度计(Thermo Scientific, USA)测定, -20℃冰箱保存备用.
1.4 水体和沉积物样品硝化微生物的实时荧光定量PCR分别以Arch-amoA F/Arch-amoA R[22]和amoA-1 F/amoA-2 R[23]为引物, 采用CFX96 Real-Time PCR System(Bio-rad, USA)实时荧光定量PCR仪测定水样与沉积物样品中的AOA和AOB amoA基因丰度. qPCR反应体系为20 μl, 包括10 μl SYBR Premix Ex Taq(Takara, Shanghai, China)、上下游引物各0.5 μl及2 μl模板, 最后加入灭菌双蒸水补足到20 μl. qPCR反应程序为初始变性温度95℃ 3.0 min;95℃ 30 s, 55℃ 20 s, 72℃ 20 s循环40次, 83℃读板;溶解曲线温度变化范围为65.0~95.0℃.以AOA和AOB amoA基因的重组质粒作为标准DNA模板, 分别以10倍梯度稀释各模板, 标准曲线浓度范围分别为3.62×102~3.62×109 copies/μl(AOA)和7.14×102~7.14×109 copies/μl(AOB). AOA和AOB的扩增效率分别为95.4 % (R2=0.997)和86.1 % (R2=0.986).水体和沉积物中的NOB均低于检测限, 因此没有做NOB的定量研究.
1.5 水体和沉积物样品AOA的PCR-DGGE、克隆测序及系统发育分析用于DGGE的AOA amoA基因PCR产物的引物为Arch-amoA F和Arch-amoA R. 50 μl PCR体系包括5 μl含有20 mmol/L Mg2+的10×HotStar-Taq buffer、2.5 μl dNTP、2.5 U的HotStar-Taq polymerase(Takara), 0.5 μmol/L的上下游引物和1 μl DNA模板, 用灭菌双蒸水补足至50 μl. AOA PCR反应条件: 94℃ 5.0 min;95℃ 30 s, 55℃ 45 s, 72℃ 45 s, 35个循环;72℃ 7 min.通过1.2 %琼脂糖凝胶电泳检测PCR扩增特异性. AOA amoA基因的DGGE分析使用6 %的聚丙烯酰胺凝胶[21], 变性梯度范围为20 % ~50 %.电泳条件为: 0.5×TAE缓冲液, 70 V电压, 60℃电泳16 h.电泳分析通过Dcode Universal Mutation Detection System(Bio-Rad)完成后, 采用SYBR Green I染料(1 :10000的稀释倍数)(Invitrogen, USA)对聚丙烯酰胺凝胶染色30 min, 使用Gel Doc XR+凝胶成像系统(Bio-rad)进行紫外成像.
在紫外灯下小心迅速地切割下DGGE胶上的典型条带, 放置于30 μl灭菌双蒸水中, 用枪尖捣碎, 4℃过夜.取1 μl洗脱出来的DNA溶液作为模板连同原始土壤总DNA进行PCR扩增, PCR扩增体系和反应条件如前所述. PCR产物参照pEASYTM-T1 Cloning Kit(TransGen, China)说明书进行克隆.每个条带随机地挑选出5个含有AOA amoA基因片段的白色克隆, 并将克隆子进行测序(Genscript, Nanjing, China).
用DNASTAR软件对测序结果进行编辑, 去除载体序列后用BLAST在GenBank数据库中搜索相似序列, 并与已发表的相关细菌和古菌的amoA功能基因序列进行多重序列比对, 使用MEGA 4.1软件以邻接法(Neighbor-Joining)构建系统发育树.
1.6 沉积物样品中AOB和NOB群落丰度分析——基因组Miseq测序本研究中, 水体和沉积物样品中AOA群落丰度采用基于amoA基因的PCR-DGGE分析获得, 由于部分沉积物样品中AOB与NOB功能基因丰度低, PCR扩增产物量无法满足DGGE分析, 故本研究采用基于16S rRNA基因的Miseq测序分析沉积物AOB与NOB的群落及丰度.对细菌16S rRNA基因V3-V4区进行PCR扩增, 引物为338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′), 每个样本的引物5′端加barcode序列, 不同样本用不同的barcode序列进行区分.每个样本3个重复, 将同一样本的PCR产物混合后用2 %琼脂糖凝胶电泳检测, 用AxyPrepDNA凝胶回收试剂盒(Axygen, USA)切胶回收PCR产物, 再用QuantiFluorTM-ST蓝色荧光定量系统(Promega, USA)进行检测定量, 按照每个样本的测序量要求, 进行相应比例的混合.利用Illumina平台进行文库构建, 由上海美吉生物公司采用Illumina Miseq方法进行测序, Miseq测序基于resample处理来分析细菌各类群相对丰度, resample序列数为24000.利用Usearch(Version 7.1)对测序数据进行处理, 按照97 %相似性对非重复序列(不含单序列)进行OTU聚类, 在聚类过程中去除嵌合体, 得到OTU的代表序列.利用DNAman将AOB和NOB主要类群序列与97 %相似度的OTUs序列进行比对[24].
1.7 数据分析采用双因素方差分析(Two-way ANOVA, Tukey多重比较)评估不同采样点与采样深度分别对太湖水体理化指标的影响和两因素对理化指标的交互影响;采用单因素方差分析(One-way ANOVA, Tukey多重比较)评估不同数据间的差异;采用独立样本T检验比较HT表层沉积物各理化指标与ET是否存在显著差异;采用Pearson相关分析评估水体与表层沉积物AOA和AOB丰度与各环境因子的关系, P < 0.05表示具有统计显著性.利用逐步回归分析进一步评估水体与表层沉积物AOA和AOB丰度及其比值与环境因子关系, 保留显著影响变量(P < 0.05).采用Bio-rad image lab 4.1软件对水体与表层沉积物中AOA amoA基因DGGE条带进行数字化分析, DGGE条带的位置和相对亮度分别表征微生物种类和数量.将DGGE图谱条带数字化结果、表层沉积物中AOB或NOB各属16S rRNA基因相对丰度与环境因子进行逐步回归分析, 筛选显著影响变量(P < 0.05).所有分析基于SPSS统计软件(SPSS, USA).
2 结果与分析 2.1 梅梁湾与湖心区水体与沉积物的理化性质梅梁湾与湖心区水样中TP、TN、NH4+-N、NO3--N和NO2--N和DO浓度均有显著差异(表 1), 其中梅梁湾水样中TP、TN、NH4+-N和NO2--N浓度显著高于湖心区水样, 而NO3--N和DO浓度显著低于湖心区水样.另外, 梅梁湾水体中不同深度水样的TP、TN和DO浓度也有显著差异, 且表层>中层>底层.但是不同湖区、不同深度的水样pH值无显著差异(表 1).双因素方差分析显示, TP、TN、NH4+-N、NO3--N和DO浓度显著受采样点和采样深度的影响.参照《湖泊(水库)富营养化评价方法及分级技术规定》, 湖泊富营养化评价参数包括Chl.a、TP、TN、CODMn和SD, 计算得出梅梁湾湖区和湖心区综合营养状态指数(TLI)分别为69和59, 分别属于中度富营养和轻度富营养区域.
| 表 1 太湖水体理化指标及双因素方差分析 Tab.1 Two-way ANOVA of physicochemical index of water in Lake Taihu |
梅梁湾表层沉积物中TOC、TP、TN和NH4+-N含量显著高于湖心区, 而NO3--N、NO2--N含量、pH和DO无显著差异(表 2).
| 表 2 太湖沉积物理化指标独立样本T检验结果* Tab.2 Independent sample-T test results of the physicochemical index of sediment in Lake Taihu |
梅梁湾湖区水体表层、中层和底层水样中AOA amoA基因的丰度分别为6.70×102、4.24×102和1.60×103 copies/ml, AOB amoA基因的丰度分别为1.96×103、5.25×102和7.64×102 copies/ml;湖心区表层、中层和底层水样中AOA amoA基因的丰度分别为1.54×103、5.48×103和7.69×103 copies/ml, AOB amoA基因的丰度分别为2.18×102、4.27×102和5.81×102 copies/ml(图 2).可见, 梅梁湾湖区水体表层、中层和底层水样中AOA amoA基因丰度分别低于湖心区, 而AOB amoA基因丰度分别高于湖心区.梅梁湾湖区的AOA/AOB丰度比显著低于湖心区, 且湖区表层、中层和底层水样中AOA/AOB丰度比逐步升高.

|
图 2 太湖水体和沉积物AOA和AOB amoA基因丰度(方框中数字表示AOA与AOB丰度比) Fig.2 Abundance of AOA and AOB expressed as amoA copy numbers per milliliter in water and per gram in sediment of Lake Taihu(The ratio of AOA to AOB were shown in the box above the chart) |
梅梁湾湖区和湖心区表层沉积物AOA amoA基因丰度分别为4.63×107和8.61×108 copies/g干土, AOB amoA基因丰度分别为6.53×107和1.32×107 copies/g干土.梅梁湾湖区的AOA/AOB丰度比低于湖心区.
逐步回归分析表明(表 3), 水体AOA amoA基因丰度与TN浓度呈显著相关(R2=0.512, P < 0.01), AOB amoA基因丰度与TP、NH4+-N浓度呈显著相关(R2=0.407, P < 0.05), AOA/AOB丰度比与TP浓度呈显著正相关, 与TN、NH4+-N和DO浓度均呈显著负相关(R2=0.852, P < 0.001);沉积物AOA amoA基因丰度与TN、TP、DO浓度均呈显著相关(R2=0.943, P < 0.001), 沉积物AOB amoA基因丰度与TN、DO浓度均呈显著相关(R2=0.720, P < 0.01), 沉积物AOA/AOB丰度比与TN浓度呈显著负相关(R2=0.370, P < 0.05).可见, 影响太湖水体和沉积物中AOA和AOB群落丰度的最主要环境因子是TN、TP与NH4+-N.
| 表 3 逐步回归分析筛选的与水体和沉积物中AOA、AOB amoA基因丰度相关的环境因子 Tab.3 Environmental factors passed through stepwise analysis to correlate with the abundance of AOA and AOB amoA gene in water and sediment |
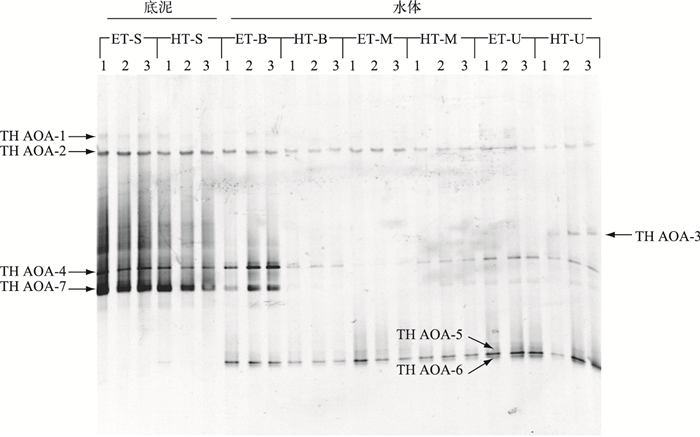
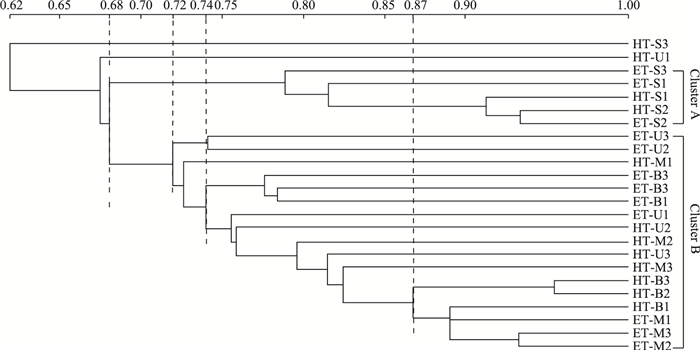
梅梁湾湖区和湖心区水样与表层沉积物样品中AOA amoA基因共有7条DGGE条带(OTU, 图 3).其中, TH AOA-1、2、3、4是水样和沉积物样品共有条带, TH AOA-5、6是水样中的特有条带, TH AOA-7是沉积物中的特有条带.聚类分析显示, 梅梁湾湖区和湖心区水样AOA的DGGE带谱聚在一簇, 两湖区沉积物AOA的DGGE带谱也聚在一簇.梅梁湾湖区和湖心区水样、表层沉积物样品中AOA群落组成基本相似(图 4).
|
图 3 太湖水体和表层沉积物氨氧化古菌amoA基因DGGE图谱 Fig.3 DGGE fingerprint of archaeal amoA gene within water and surface sediments in Lake Taihu |
|
图 4 太湖水体和表层沉积物AOA amoA基因DGGE指纹图谱聚类分析 Fig.4 Cluster analysis of DGGE fingerprint of archaeal amoA gene of water and surface sediments in Lake Taihu |
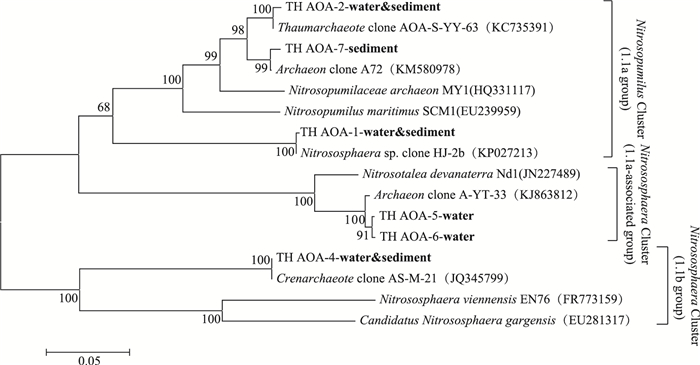
克隆测序及系统发育分析表明(图 5), AOA amoA基因序列可分为3类, TH AOA-1、2和7属于Nitrosopumilis(1.1a group), 与海洋水体和沉积物的AOA亲缘关系较近;TH AOA-4属于Nitrosophaera(1.1b group), 与土壤中的AOA亲缘关系较近.水样中特有序列TH AOA-5和6属于Nitrosotalea(1.1a-associated group), 多见于酸性水稻土壤中, 且与酸性土壤硝化古菌Nitrosotalea devanaterra具有最近的亲缘关系.
|
图 5 太湖水体和表层沉积物氨氧化古菌amoA基因系统发育分析 Fig.5 Phylogenetic tree showing the relationship between the archeal amoA gene sequences obtained from DGGE bands |
基于16S rRNA基因Miseq高通量测序结果表明, 太湖表层沉积物样品中AOB群落主要包括亚硝化螺旋菌(Nitrosospira)和亚硝化单胞菌(Nitrosomonas)两大属, 其中梅梁湾湖区表层沉积物中AOB群落以亚硝化螺旋菌为主, 其相对丰度为97.54 %;湖心区表层沉积物中亚硝化螺旋菌与亚硝化单胞菌相对丰度分别为48.57 %和51.43 % (图 6).富营养化水平不同的梅梁湾湖区和湖心区表层沉积物中AOB群落丰度有明显差异.
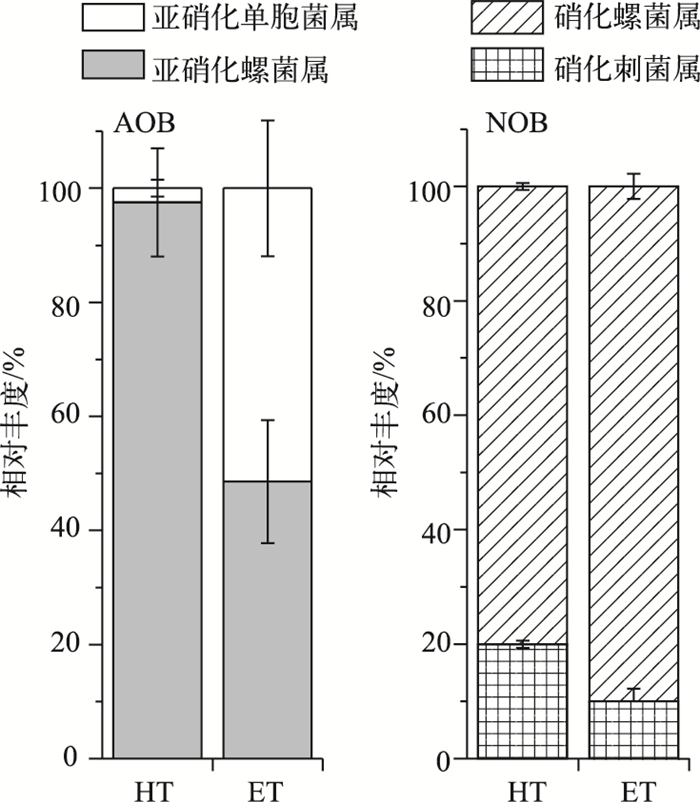
|
图 6 采样点HT和ET沉积物中氨氧化细菌及亚硝酸盐氧化菌16S rRNA基因相对丰度 Fig.6 Relative abundance of ammonia-oxidizing and nitrite-oxidizing bacteria 16S rRNA gene sequences of the sediments in HT and ET sites |
太湖表层沉积物样品中NOB群落主要包括硝化螺菌(Nitrospira)和硝化刺菌(Nitrospina)两大属, 梅梁湾湖区与湖心区表层沉积物中均以硝化螺菌为主, 2个湖区硝化螺菌的相对丰度分别为80.0 %和90.0 % (图 6).富营养化水平不同的梅梁湾湖区和湖心区表层沉积物中NOB群落丰度有明显差异.
2.5 AOA、AOB与NOB群落丰度与环境因子相关性分析HT与ET采样点水体和表层沉积物AOA及表层沉积物AOB与NOB群落丰度与环境因子的关系如表 4.逐步回归分析显示, 水体与沉积物共有条带AOA-1、2和沉积物特有条带AOA-7所属的Nitrosopumilis(1.1a group)丰度主要与NO3--N浓度呈显著正相关, 水体特有条带AOA-5、6所属的Nitrosotalea(1.1a-associated group)丰度主要与pH、DO浓度呈显著正相关.表层沉积物中亚硝化单胞菌(Nitrosomonas)、亚硝化螺菌(Nitrosospira)丰度与TN浓度呈显著相关, 硝化螺菌(Nitrospira)、硝化刺菌(Nitropina)丰度均与NO2--N浓度呈显著相关.
| 表 4 AOA、AOB与NOB群落丰度与环境因子的逐步回归分析 Tab.4 Stepwise regression analysis between AOA, AOB and NOB community composition and the environmental factors |
本研究多角度地研究中度富营养化的太湖梅梁湾湖区和轻度富营养化的湖心区水体与表层沉积物中氨氧化微生物、亚硝酸氧化微生物的分布、群落组成和丰度等.梅梁湾湖区与湖心区水样中AOA群落组成基本相似, 两湖区表层沉积物样品中AOA群落组成亦基本相似, 水体中AOA群落组成与表层沉积物中AOA群落组成有差异;两湖区表层沉积物中AOB与NOB群落丰度均有明显差异.
太湖2个湖区表层沉积物的AOA amoA序列属于海洋古菌类群1.1a group和土壤古菌类群1.1b group, 证实了从太湖沉积物中获得的大部分AOA amoA序列与土壤和海洋环境中的AOA amoA序列有关[12, 14];水体特有AOA amoA序列属于水稻土壤类群1.1a-associated group, 此可能与土壤径流有关.逐步回归分析结果表明, Nitrosopumilis(1.1a group)丰度主要与NO3--N呈显著正相关, 说明Nitrosopumilis可以促进硝酸盐的积累;Nitrosotalea(1.1a-associated group)丰度主要与pH、DO呈显著正相关, DO作为氨氧化反应底物是影响水体氨氧化微生物群落组成及丰度的重要因素;pH能通过影响氨氧化反应底物的化学形态、浓度与有效性来影响含amoA基因微生物的群落组成及丰度[25].
太湖梅梁湾湖区和湖心区表层沉积物AOB群落主要包括亚硝化螺菌和亚硝化单胞菌两大属.亚硝化螺菌和亚硝化单胞菌是淡水湖泊生态系统最常见的2种AOB[12, 26-28].中度富营养化的梅梁湾湖区表层沉积物AOB优势菌为亚硝化螺菌, Xing等[29]也有相似研究结果.梅梁湾湖区表层沉积物中亚硝化螺菌的相对丰度远高于湖心区, 随着营养化水平的降低, 亚硝化螺菌相对丰度下降; 在轻度营养化的湖心区表层沉积物中, 亚硝化螺菌和亚硝化单胞菌丰度基本接近, 亚硝化螺菌可能比亚硝化单胞菌更适应富营养水平较高的环境.逐步回归分析结果表明, 太湖表层沉积物中TN含量显著影响AOB群落丰度.沉积物中微生物可以促进有机氮的矿化, 矿化产生的氨是氨氧化过程的底物和氨氧化微生物的主要能量来源, 因此, TN浓度可以提高氨氧化微生物的活性[14], 从而影响AOA或AOB的群落组成与丰度[30].
太湖梅梁湾湖区和湖心区表层沉积物中NOB主要包括硝化螺菌和硝化刺菌两大属, 在太湖中发现的硝化刺菌是淡水湖泊中比较少见的NOB.硝化刺菌多见于海洋环境, 适宜在缺氧的深海环境中生长[11, 13, 31], 太湖中特有的硝化刺菌可能是由于太湖地区历史上经历过多次海陆交替而留存[32].在湖心区表层沉积物中发现54.35 %的硝化刺菌为Candidatus Nitrotoga arctica DQ839562.1(BLAST identity参数为95 %), 该菌能适应冷环境并可在10~17℃的环境中被富集[33].水体富营养化水平能够影响表层沉积物中NOB的群落丰度, 湖心区表层沉积物中硝化螺菌的比例高于梅梁湾湖区.逐步分析结果表明, 作为NOB催化的反应底物NO2--N, 其含量是影响NOB群落丰度的主要因素.
3.2 梅梁湾与湖心区水体和沉积物中AOA与AOB丰度分布的环境影响因子研究表明, 淡水水体和沉积物中氨氧化微生物丰度和分布通常受NH4+-N、TN、无机盐、DO、pH、温度等因素的影响[11, 14-15, 19].本研究显示, 水体AOA丰度与TN浓度呈显著相关, AOB丰度与TP、NH4+-N浓度均呈显著相关;表层沉积物AOA丰度与TN、TP和DO浓度均呈显著相关, AOB丰度与TN、DO浓度呈显著相关.根据文献[12, 34]报道, 淡水湖泊沉积物中的TP含量是AOA丰度的显著影响因子, 可能是由于AOA基因组含有P相关的代谢基因, 如Nitrosospira含有催化合成6-磷酸果糖的葡萄异构酶的所有编码基因[35].
影响水体AOA/AOB丰度比的主要因素有TN、NH4+-N、TP和DO, 2个湖区水样从上至下的TN、NH4+-N、TP和DO值都呈下降趋势, 表明AOB更适合在高氮、高磷的富营养环境下生长, 而AOA更适宜在相对低氮、低磷的富营养水平低或贫营养环境中存活[34, 36].在氮含量较少的情况下, AOA在与其他微生物竞争底物NH4+-N时占有绝对的优势.因此, 在NH3浓度极低的贫营养水体中AOA丰度远高于AOB. AOA/AOB丰度比在水体中的垂直分布差异还与它们对氧气的需求有关, AOA广泛地分布于海洋、土壤、沉积物环境中好氧到缺氧的环境中[21], 它们更适应缺氧的环境.太湖是一个浅层湖泊, 它的水体和表层沉积物有着比较充分的交换作用, 氧气能够通过交换作用从水中传递到表层沉积物中[34].而太湖水体从上到下, DO值不断下降, AOA在DO值较低的底层水体中更具优势.
梅梁湾湖区和湖心区表层沉积物的AOA/AOB的比率分别为0.71和65.26, 梅梁湾湖区表层沉积物中AOB更具优势, 湖心区则相反, 与水体中AOA与AOB的分布趋势基本一致.此结果与Hou等[6]的相似, 但与Wu等[12]和Zeng等[14]的结果有差异, 可能是由于采样时间与季节不同引起的各湖区富营养程度的差异而造成的.研究同时表明, 影响表层沉积物AOA amoA基因丰度的主要因素有TN、TP和DO, 影响表层沉积物AOB amoA基因丰度的主要因素有TN和DO, 影响表层沉积物AOA/AOB丰度比的主要因素有TN, 说明氮、磷和溶解氧的含量是影响沉积物中氨氧化微生物丰度的主要因素, 此结果与文献[8, 11, 15]一致.
4 结论典型富营养化指标(TN、TP、NH4+-N、NO3--N和NO2--N等)是影响太湖梅梁湾和湖心区水体和沉积物中AOA或AOB丰度以及硝化微生物群落丰度的重要因素.中度富营养化的梅梁湾湖区表层、中层水样与表层沉积物样品的AOB丰度高于AOA, 轻度富营养化的湖心区水样与表层沉积物样品AOA丰度高于AOB.梅梁湾湖区和湖心区水样中AOA群落组成基本相似, 表层沉积物样品中AOA群落组成基本相似, 水体中AOA群落组成与表层沉积物中AOA群落组成有差异;梅梁湾湖区AOB优势菌为亚硝化螺旋菌, 湖心区的亚硝化螺旋菌与亚硝化单胞菌的丰度基本相同;两湖区NOB均以硝化螺菌为主.研究表明富营养化程度显著影响太湖梅梁湾和湖心区水体或沉积物中硝化微生物分布和群落丰度, 有助于全面了解太湖生态系统氮素循环及相关功能微生物.
| [1] |
Canfield DE, Glazer AN, Falkowski PG. The evolution and future of Earth's nitrogen cycle. Science, 2010, 330(6001): 192-196. DOI:10.1126/science.1186120 |
| [2] |
Galloway JN, Townsend AR, Erisman JW et al. Transformation of the nitrogen cycle:Recent trends, questions and potential solutions. Science, 2008, 320(5878): 889-892. DOI:10.1126/science.1136674 |
| [3] |
Sun Xiaojing, Qin Boqiang, Zhu Guangwei. Release of colloidal phosphorus, nitrogen and organic carbon in the course of dying and decomposing of cyanobacteria. China Environmental Science, 2007, 27(3): 341-345. [孙小静, 秦伯强, 朱广伟. 蓝藻死亡分解过程中胶体态磷、氮、有机碳的释放. 中国环境科学, 2007, 27(3): 341-345.] |
| [4] |
Zhu Guangwei, Qin Boqiang, Gao Guang et al. Accumulation characteristics of heavy metals in the sediments of Lake Taihu. J Lake Sci, 2005, 17(2): 143-150. [朱广伟, 秦伯强, 高光等. 太湖近代沉积物中重金属元素的累积. 湖泊科学, 2005, 17(2): 143-150. DOI:10.18307/2005.0209] |
| [5] |
Qin Boqiang, Hu Weiping, Chen Weimin et al. Evolution process and mechanism of aquatic environment in Taihu. Beijing: Science Press, 2004. [秦伯强, 胡维平, 陈伟民等. 太湖水环境演化过程与机理. 北京: 科学出版社, 2004.]
|
| [6] |
Hou J, Song CL, Cao XY et al. Shifts between ammonia-oxidizing bacteria and archaea in relation to nitrification potential across trophic gradients in two large Chinese lakes (Lake Taihu and Lake Chaohu). Water Research, 2013, 47(7): 2285-2296. DOI:10.1016/j.watres.2013.01.042 |
| [7] |
Nicol GW, Leininger S, Schleper C et al. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia-oxidizing archaea and bacteria. Environ Microbiol, 2008, 10: 2966-2978. DOI:10.1111/emi.2008.10.issue-11 |
| [8] |
Zhao DY, Zeng J, Wan WH et al. Vertical distribution of ammonia-oxidizing archaea and bacteria in sediments of a eutrophic lake. Curr Microbiol, 2013, 67: 327-332. DOI:10.1007/s00284-013-0369-7 |
| [9] |
Sorokin DY, Lucker S, Vejmelkova D et al. Nitrification expanded:Discovery, physiology and genomics of a nitrite-oxidizing bacterium from the phylum Chloroflexi. ISME, 2012, 6: 2245-2256. DOI:10.1038/ismej.2012.70 |
| [10] |
Holger D, Elena VL, Petra P et al. Complete nitrification by Nitrospira bacteria. Nature, 2015, 528: 504-509. |
| [11] |
Wang Chao, Shan Baoqing. The distribution of aerobic ammonia oxidizing microorganisms in Ziya River, Haihe Basin. Acta Scientiae Circumstantiae, 2012, 32(12): 2943-2950. [王超, 单保庆. 子牙河水系水和沉积物好氧氨氧化微生物分布特征. 环境科学学报, 2012, 32(12): 2943-2950.] |
| [12] |
Wu YC, Xiang Y, Wang JJ et al. Heterogeneity of archaeal and bacterial ammonia oxidizing communities in Lake Taihu, China. Environ Microbiol Rep, 2010, 2: 569-576. DOI:10.1111/emi4.2010.2.issue-4 |
| [13] |
Liu B, Li YM, Zhang JP et al. Abundance and diversity of ammonia-oxidizing microorganisms in the sediments of Jinshan Lake. Curr Microbiol, 2014, 69: 751-757. DOI:10.1007/s00284-014-0646-0 |
| [14] |
Zeng J, Zhao DY, Huang R et al. Abundance and community compositon of ammonia-oxidizing archea and bacteria in two different zones of Lake Taihu. Canadian Journal of Microbiology, 2012, 58(8): 1018-1026. DOI:10.1139/w2012-078 |
| [15] |
Liu Y, Zhang JX, Zhao L et al. Distribution of sediment ammonia-oxidizing microorganisms in plateau freshwater lakes. Appl Microbiol Biotechnol, 2015, 99: 4435-4444. DOI:10.1007/s00253-014-6341-z |
| [16] |
Chen Guoyuan, Huang Xiaoming. Distribution and role of nitrifying bacteria in the sediments of Xihu Lake in Quanzhou. Microbiology China, 2011, 38(11): 1632-1638. [陈国元, 黄晓鸣. 泉州西湖沉积物中硝化细菌的分布及其作用. 微生物学通报, 2011, 38(11): 1632-1638.] |
| [17] |
Attard E, Poly F, Commeaux C et al. Shifts between Nitrospira-and Nitrobacter-like nitrite oxidizers underlie the response of soil potential nitrite oxidation to changes in tillage practices. Environmental Microbiology, 2010(12): 315-326. |
| [18] |
Manser R, Gujer W, Siegrist H. Decay processes of nitrifying bacteria in biological wastewater treatment systems. Water Research, 2006, 12(40): 2416-2426. |
| [19] |
Gutierrez T, Singleton DR, Berry D et al. Hydrocarbon-degrading bacteria enriched by the Deepwater Horizon oil spill identified by cultivation and DNA-SIP. International Society for Microbial Ecology, 2013, 7: 2091-2104. |
| [20] |
Jin Xiangcan, Tu Qingying eds. Investigation specification for limnology eutrophication. Second Edition. Beijing: Chinese Evironmental Science Press, 1990. [金相灿, 屠清瑛. 湖泊富营养化调查规范. 第二版. 北京: 中国环境科学出版社, 1990.]
|
| [21] |
Wang XL, Han C, Zhang JB et al. Long-term fertilization effects on active ammonia oxidizers in an acid upland soil in China. Soil Biology & Biochemistry, 2015, 84: 28-37. |
| [22] |
Francis CA, Roberts KJ, Beman JM et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean. Proc Natl Acad Sci USA, 2005, 102: 14683-14688. DOI:10.1073/pnas.0506625102 |
| [23] |
Rotthauwe JH, Witzel KP, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. |
| [24] |
Chen CJ, Sun FQ, Zhang HQ et al. Evaluation of COD effect on anammox process and microbial communities in the anaerobic baffled reactor (ABR). Bioresource Technology, 2016, 216: 571-578. DOI:10.1016/j.biortech.2016.05.115 |
| [25] |
Nicol GW, Leininger S, Schleper C et al. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria. Environmental Microbiology, 2008, 10(11): 2966-2978. DOI:10.1111/emi.2008.10.issue-11 |
| [26] |
Chen GY, Qiu SL, Zhou YY. Diversity and abundance of ammonia-oxidizing bacteria in eutrophic and oligotrophic basins of a shallow Chinese lake (Lake Donghu). Research in Microbiology, 2009, 160(3): 173-178. DOI:10.1016/j.resmic.2009.01.003 |
| [27] |
Monique JMdB, Arjen GCLS, George AK et al. Shifts in the dominant populations of ammonia-oxidizing betasubclass Proteobacteria along the eutrophic Schelde estuary. Aquatic Microbial Ecology, 2001, 23(3): 225-236. |
| [28] |
Koops HP, Pommerening-Röser A. Distribution and ecophysiology of the nitrifying bacteria emphasizing cultured species. FEMS Microbiology Ecology, 2001, 37(1): 1-9. DOI:10.1111/fem.2001.37.issue-1 |
| [29] |
Xing P, Kong FX, Chen KN et al. Ammonia-oxidizing bacteria community composition at the root zones of aquatic plants after ecological restoration. Environment Science, 2008, 29(8): 2154-2159. |
| [30] |
Zheng YL, Hou LJ, Newell S et al. Community dynamics and activity of ammonia-oxidizing prokaryotes in intertidal sediments of the Yangtze Estuary. Applied and Environmental Microbiology, 2014, 80(1): 408-419. DOI:10.1128/AEM.03035-13 |
| [31] |
Eva S, Sabine K, Thilo W et al. Characterization of a new marine nitrite oxidizing bacterium, Nitrospina watsonii sp. nov., a member of the newly proposed phylum "Nitrospinae". Systematic and Applied Microbiology, 2014, 37(3): 170-176. DOI:10.1016/j.syapm.2013.12.005 |
| [32] |
Wang Suming, Dou Hongshen. Lakes of China. Beijing: Science Press, 1998, 261-268. [王苏民, 窦鸿身. 中国湖泊志. 北京: 科学出版社, 1998, 261-268.]
|
| [33] |
Christner BC, Priscu JC, Amanda M. A microbial ecosystem beneath the West Antarctic ice sheet. Nature, 2014, 512: 310-319. DOI:10.1038/nature13667 |
| [34] |
Rippey B, Jewson DH. The rates of sediment-water exchange of oxygen and sediment bioturbation in Lough Neagh, Northern Ireland. Hydrobiologia, 1982, 91/92(1): 377-382. DOI:10.1007/BF02391953 |
| [35] |
Spang A, Poehlein A, Offre P et al. The genome of the ammonia-oxidizing Candidatus Nitrososphaera gargensis:Insights into metabolic versatility and environmental adaptations. Environmental Microbiology, 2012, 14(12): 3122-3145. DOI:10.1111/emi.2012.14.issue-12 |
| [36] |
Yang YY, Zhang JX, Zhao Q et al. Sediment ammonia-oxidizing microorganisms in two plateau freshwater lakes at different trophic states. Microbial Ecology, 2016, 71: 257-265. DOI:10.1007/s00248-015-0642-3 |
 2017, Vol. 29
2017, Vol. 29 

