(2: 中国科学院大学中丹学院, 北京 100049)
(3: 中国科学院大学, 北京 100049)
(2: Sino-Danish College, University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
(3: University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
湖泊是陆地生态系统重要的生态类型之一,是陆地水圈的重要组成部分[1-2].在湖泊生态系统中,生物和环境两者紧密联系、相互作用,在区域乃至全球尺度上的元素循环中发挥着重要作用.微生物是湖泊生态系统中物质循环和能量流动的重要参与者,在维持生态系统平衡和驱动元素循环中起着关键性作用[3].湖泊微生物的研究,对于揭示湖泊生态系统的元素循环过程及其对环境变化的响应机制,以及深入了解湖泊生态系统结构和功能有着重要意义[4].
在传统的微生物相关研究中,微生物的分离与培养扮演着至关重要的角色[5-6].但是,由于对自然界中微生物生长所需营养物质以及微生物之间普遍存在的复杂共生关系认识有限[7],自然界中绝大部分微生物在实验室中难以被培养,尤其是淡水和海洋中的浮游微生物,其可培养率分别为0.25 %和0.001 % ~0.1 % [8].因此,湖泊中的绝大多数微生物还未被人们所认知,对其功能的认识更为匮乏.
在过去的20年中,快速发展的测序技术和计算能力已经为微生物生态学领域带来革命性的影响.不依赖培养的微生物研究技术方法不断建立,宏基因组学技术就是其中发展最快、应用最广泛的方法之一[5, 9]. 1998年,Handelsman首次提出了宏基因组(Metagenome)的概念,即环境样本中全部微生物基因组的总和,宏基因组学(Metagenomics)是将环境中全部微生物的遗传信息看作一个整体,自上而下地研究微生物与自然环境或其他生物体之间关系的一种方法[9].这里需要说明的是,扩增子测序(是对特定长度的PCR产物或捕获的片段进行测序,分析序列中的变异和丰度,主要用于研究环境微生物多样性及群落组成差异)尽管也被归入宏基因组学方法,但其不在本文讨论的范畴.宏基因组学方法一定程度上突破了水体微生物难以培养的困境,而且通过与生物信息学的有机结合,在揭示水体微生物之间、微生物与环境之间相互作用的规律中发挥了巨大的支撑作用,有效地拓展了湖泊微生物的研究思路与方法,为从群落水平上全面认识湖泊微生物的生态特征和功能开辟了新的途径[10-11].
目前,宏基因组学作为迄今为止最全面地了解微生物群落特征、最大限度地挖掘微生物资源的一种方法,已经成为了国际上微生物生态学主要的研究手段.随着高通量测序技术的不断发展,测序成本不断下降,宏基因组学技术将会越来越多地应用于湖泊微生物的相关研究.本文通过文献计量分析和数据库检索方法展示了宏基因组学在湖泊微生物生态学中的应用现状,重点介绍了目前的研究热点问题;在方法学部分着重介绍了湖泊宏基因组学生物信息学分析中关键步骤——数据分装(Binning)的发展趋势;文末展望了未来湖泊微生物宏基因组学研究的发展趋势和研究重点.
1 湖泊宏基因组研究文献计量分析 1.1 国际研究文献计量分析本文研究数据来源于Web of Science (WOS)中的科学引文索引扩展版(Science Citation Index Expanded, 简称SCI-E),分别以主题词:lake & marine & ocean & soil & atmosphere & air & metagenom*对SCI-E数据库时间范围为2008-2018年的文献进行检索.检索时间为2018年11月20日,检索文献类型界定为“论文”和“综述”,不包括会议录文献、会议摘要、书评、信函、社论材料等.共检索到文献3551篇,其中涉及湖泊文献282篇,海洋1474篇,土壤1125篇,大气及其他环境670篇,湖泊微生物相关研究仅占到检索文献总数的7.9 %.目前,宏基因组学研究方法在海洋和土壤微生物生态学研究中已经受到普遍关注,而在湖泊生态系统中的应用仍处于逐年增加的阶段.湖泊微生物宏基因组学相关文章从2008年的6篇增至2018年的超过50篇. 282篇有关湖泊微生物宏基因组的研究论文共发表在104种SCI-E期刊上,其中34篇发表在8种自然指数收录期刊上,占全部检索论文的12.1 %.国际微生物生态学会会刊The ISME Journal发表湖泊宏基因组相关研究论文23篇,位列8种自然指数期刊第一.
以检索获得的282篇文献为研究对象,对数据合并、去重等清洗后进行各指标定量分析,同时结合文献阅读和湖泊生态学领域专家的建议,近10年来,湖泊微生物宏基因组学研究大致可以归纳为以下几个主要方向:1)探索各种类型湖泊中的未知微生物结构和功能,不仅提供物种存在的基因组学证据,还可以通过基因组代谢特征分析,直接预测未知微生物在生态系统中的功能;2)从微生物群落水平,揭示湖泊生态系统中物质循环关键代谢途径和其主要微生物功能类群;3)通过深度测序重构微生物基因组草图,开展微生物在湖泊环境的适应性进化研究、揭示演化过程和规律.
1.2 湖泊宏基因组数据产出分析在文献计量分析学分析基础上,本研究继续对上传到美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)数据库的Sequence Read Archive(SRA)进行检索和信息提取.将研究过程中产出的数据上传到公共数据库,是目前主流SCI-E期刊对于论文投稿的基本要求.由于数据库中有大量尚未发表的研究工作提交的宏基因组数据,因此对数据库已有信息的挖掘和整合有助于更为全面地掌握湖泊微生物宏基因组学研究动态.在https://www.ncbi.nlm.nih.gov/sra/下使用“lake”作为关键词进行检索,共获得57943条记录(检索时间为2019年4月18日),其中DNA来源数据53993条.通过设定筛选条件,可以对满足条件的数据集进行深度分析.
本研究重点分析了湖泊水体宏基因组全球数据分布的情况.通过确定分析类型(Assay_Type=WGA: whole genome amplification & WGS: whole genome sequencing & other),设定数据量阈值(MBytes >100Mb)以及筛选测序方法后,获得SRA数据1941条.在SRA对应的测序项目(Bioproject)和样品(Biosample)信息中提取湖泊经纬度、样品数量和数据量,进一步制作湖泊宏基因组数据全球分布图(图 1).根据数据来源湖泊归属国家和地区进行排序,美国、非洲、南极洲、加拿大和中国是数据量排名前5位的区域.世界储水量和深度均排名第二的坦噶尼喀湖是目前储备宏基因组数据最多的湖泊(0.62 TBytes).太湖是我国目前微生物宏基因组学数据量最集中的湖泊,共有样品记录26个,数据合计0.11 TBytes.本文作者在抚仙湖开展的宏基因组测序(PRJNA531348)是目前我国湖泊单样品测序深度最大的数据集,5个样品的数据量与太湖全部的数据量相当.
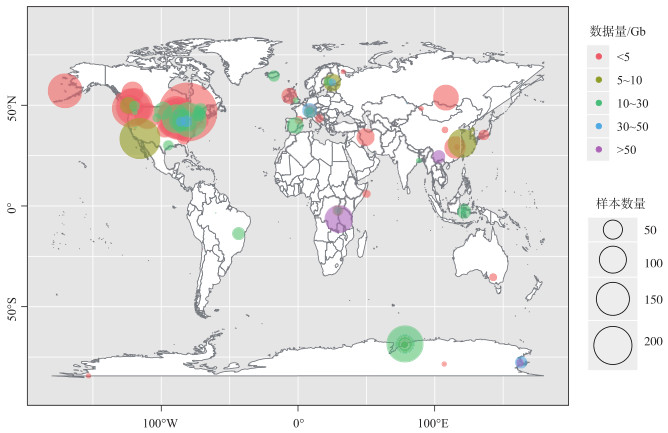
|
图 1 湖泊宏基因组数据全球分布概况 Fig.1 Global contribution of lake metagenomic raw data in NCBI database |
随着湖泊微生物研究的深入,仅仅获取湖泊微生物群落结构信息,已经远远不能满足对湖泊生态系统认识的需求.宏基因组学利用免培养手段,对环境样品中的全部基因组信息进行分析,可以全面、真实地获取湖泊微生物群落的功能,包括生理生化、物质代谢过程和环境适应机制等等;基于功能基因和代谢通路分析,可以对微生物在湖泊关键物质,例如碳、氮、硫等元素循环中发挥的作用有一个更为全面的认识.开展宏基因组学研究,有助于不断丰富和提升对湖泊微生物原位代谢特征的认识,在此基础上设计更有针对性的培养基和培养方法,扩大可分离培养微生物的范围,实现非培养与培养研究手段的有机融合.
2.1 获得湖泊不可培养微生物基因组信息随着组学和测序技术地不断发展,免培养研究手段为获取湖泊中未培养微生物的基因组信息提供了可能[12].宏基因组学通过Binning手段,将整个样本基因组集分装成一个个单一物种的基因组子集,从而可以获取较多的单一菌株的微生物基因组信息,即metagenome-assembled genomes(MAGs). 2011年,Hess首次利用宏基因组学Binning从268 Gb的牛瘤胃样品宏基因组数据中成功获取了15个高质量的未培养微生物的基因组序列,并用单细胞全基因组测序方法加以验证[13].自此,宏基因组学Binning逐渐成为了微生物宏基因组学研究的常用手段. 2017年,Bowers RM联合54位活跃在宏基因组研究前沿的学者,在《Nature Biotechnology》杂志发表论文提出MAGs质量划分标准体系[14](表 1).在获得MAGs的基础上,利用CheckM[15]等软件依据相应算法和通用标记基因集对MAGs的完整度、污染度等进行评估,确保MAGs的可靠性以及相关分析的科学性.随着测序质量和深度地不断提高,高质量MAGs可以提供的基因组信息已经逐渐接近单基因组的水平.
| 表 1 基因组草图质量标准(MAGs) Tab. 1 Genome sketch standards(MAGs) |
近几年来,Binning方法在揭示湖泊微生物组成和功能研究中发挥着重要作用. Vavourakis等在高盐湖泊的宏基因组样品中,利用Binning手段获得了分属于细菌、古菌等45个门的871个MAGs(其中154个MAGs达到高质量MAGs标准,717个满足中等质量MAGs标准),并且对所有MAGs进行了系统发育分析和碳、氮、硫循环相关功能基因分析.结果显示包括Actinobacteria在内的至少4个门(phylum)中,存在与湖泊碳固定和异化相关的未知微生物[16]. Arora-Williams在Upper Mystic湖的宏基因组数据中利用Binning手段获得了87个MAGs(完整度大于70 %,污染度小于10 %),并采用功能基因、16S rRNA和MAGs信息三者相结合的方法确定了在一系列生物化学过程,例如铁氧化和还原、硫氧化和还原、甲烷氧化、甲醇氧化、氨氧化、反硝化,发挥作用的微生物,并发现部分微生物可以在氧化甲烷和硫化物的过程中耦合硝酸盐还原过程[17]. Cabello-Yeves将贝加尔湖宏基因组数据Binning结果进行系统发育分析和功能基因分析,发现尽管湖泊被厚冰或雪覆盖,光合作用在湖泊微生物中仍普遍存在,且发现淡水中的SAR11亚型Ⅰ/Ⅱ与贝加尔湖中的Pelagibacter ubique菌株极为相似[18].针对MAGs功能挖掘,填补了湖泊中不可培养微生物的物种信息及其在湖泊中所扮演的功能角色信息.
2.2 获取湖泊微生物的群落功能特征澳大利亚新南威尔士大学Ricardo Cavicchioli教授及其合作研究团队,运用宏基因组手段长期开展南极洲低温高盐湖泊微生物生态学研究,对揭示极端湖泊生态系统中微生物在物质循环和能量流动中的作用做出了重要贡献. Organic湖是一个由海水形成的高盐浅水湖泊,且在湖泊水体中存在有文献记载以来的自然水体中最高浓度的二甲基硫化物[19].研究人员通过在宏基因组数据中查找代谢过程关键功能基因,重构代谢通路的方法,揭示了微生物对二甲基巯基丙酸的解离、碳混养(光能异养和无机质化能异养)和氮的循环矿化可能是微生物对Organic湖营养限制等特殊环境条件的适应机制.有着14 ka发育历史的Ace湖,是南极最典型的半混合型(meromectic)湖泊,绿硫细菌在Ace湖中占主导地位,执行非常活跃的硫元素形态转化过程,主要包括同化硫酸盐还原、异化硫酸盐还原和硫氧化等.在湖泊无光处绿硫细菌主要驱动硫酸盐还原过程,而在湖泊有光处绿硫细菌主要驱动硫化氢氧化为硫酸根的过程.研究还表明,Ace湖生态系统的稳定程度主要取决于极地光周期对绿硫细菌在初级生产和养分循环中主导作用的影响,以及噬菌体对于微生物群落内各成员间合作的影响[20].
“蓝藻界”(cyanosphere)内蓝藻与异养细菌之间的相互作用研究,为揭示蓝藻水华暴发机制提供了线索.淡水湖泊水体富营养化以及随之而来的蓝藻水华暴发已经成为世界范围关注的重大水环境问题.通过宏基因组学研究不仅揭示了蓝藻物种组成的变化伴随着蓝藻界内异养细菌群落的显著变化[21],而且还获得了蓝藻与异养细菌之间相互作用的证据.通过对惠氏微囊藻T100及其附生细菌群落进行功能分析发现,附生细菌不仅为微囊藻提供必须的维生素,还能够消除周围环境中对微囊藻生长不利的因素,从而使微囊藻在条件适宜时迅速形成水华,同时产生更多的次级代谢物供附生细菌生长,这种互利关系有助于微囊藻和附生细菌在复杂的水体环境中更好地生存[22].此外,研究发现尽管微囊藻本身无法固氮,但是其与附生微生物作为一个整体可以进行固氮,这可能成为非固氮蓝藻在氮相对缺乏状态下获得竞争优势的重要原因[23].
2.3 生态基因组学在湖泊研究中的发展新兴的生态基因组学弥补了遗传学在实验室和自然环境研究之间的空隙:当前的实验室遗传研究主要集中在认识基本的细胞发育过程,而自然遗传更注重在遗传适应性分析和生物体相互作用层面开展系统研究.研究人员分析了两个淡水湖Mendota湖和Trout Bog湖的总计184个宏基因组样品,通过Binning手段获得了19个属于Verrucomicrobia的MAGs.研究中对MAGs所包含的糖苷水解酶类相关基因进行了分析,结果显示Verrucomicrobia在淡水湖泊糖降解中发挥重要作用;两个湖泊糖苷水解酶基因丰度和功能存在显著差异,反映了微生物对湖泊内、外源有机碳组成差异的适应特征[24]. Cuadrat等利用Anti-SMASH和NAPDOS相应流程筛选MAGs中的次级代谢基因,在121个MAGs中鉴定出243个次级代谢物基因簇,且发现18个非核糖体肽合酶(NRPS)、19个聚酮合酶(PKS)和3个杂合PKS/NRPS簇,揭示了在湖泊中挖掘和研究次级代谢相关功能基因的潜力[25]. Mehrshad等在3个淡水湖泊的57个宏基因组样品中利用Binning手段获取了属于Chloroflexi的53个MAGs,并对其系统发育关系和进化进行了分析,结果表明盐度是海洋和淡水环境中Chloroflexi群落组成的主要影响因素[26].值得注意的是,Andrei等在分别位于捷克和瑞士的两个淡水湖中利用宏基因组学Binning手段获得了60个属于Planctomycetes的MAGs,并进行了后续的微生物进化、系统发育和基因组功能信息相关的一系列分析[27],首次提出沉积物或土壤中的Planctomycetes成功过渡到水生环境,且在淡水环境中产生了新的特定进化枝.引入生态基因组学的理念,开展微生物对湖泊生境的适应性进化研究、揭示演化过程和规律是湖泊微生物生态学发展的新方向.
3 宏基因组生物信息分析流程数据分析是宏基因组学研究的基础,由于数据信息量和复杂程度远远高于扩增子测序,因此在大规模的数据中获取有效信息是宏基因组研究的目标同时也是挑战.目前Binning成为宏基因组生物信息分析流程中发展最快、创新最多的核心技术,本节在简要介绍测序技术发展和宏基因组数据分析基本流程的基础上,重点介绍了Binning策略的发展和应用情况.
3.1 高通量测序技术和宏基因组生物信息学分析流程宏基因组学研究与高通量测序技术的发展密不可分.高通量测序技术又称“下一代”测序技术,以能一次并行对几十万到几百万条DNA分子进行序列测定和一般读长较短等为标志.目前高通量测序以Illumina公司提供的平台为主,也是湖泊微生物宏基因组学研究中应用最广泛的测序技术.第二代测序技术自身存在的局限性,如序列读长短(< 500 bp)、样品准备过程繁琐以及基因表达等相关分析准确性低等[28],催生测序技术的革新.以单分子实时测序[29]和纳米孔单分子技术[30]为典型代表的第三代测序技术显著提高序列读长(平均10~15 kb),但是较高的错误率(可以达到15 %)仍然影响组装质量.尽管通过提高测序覆盖度可以有效改善第三代测序的准确性,但测序成本和所需时间远远超过第二代测序,导致第三代测序在宏基因组学研究中并未得到广泛应用.目前,采用第二代测序和第三代测序相结合,通过高质量的二代测序短片段来校正第三代测序产生的错误碱基,可以有效改善细菌等小基因组测序的准确性(错误率低于1 %).由于宏基因组测序所要求的测序覆盖度较大,数据量庞大,这种混合组装的模式目前很难应用于宏基因组学研究中.本文以Illumina测序平台获得原始测序数据为例,开展宏基因组学生物信息学分析主要环节如图 2所示.

|
图 2 宏基因组学生物信息学分析流程 Fig.2 Metagenomics bioinformatics analysis flow based on Illumina sequencing |
宏基因组学的序列分析重要的一步就是测序片段的Binning,其准确性直接影响宏基因组学研究的精度和效率.宏基因组学Binning是将样本的整体序列集(reads或contigs等)分离成若干个不同个体的子序列集(Bins),即将同一物种的序列聚到一起,Bins中序列就是这个物种基因组的部分片段.根据基于聚类的对象不同,可以将Binning分为3类:reads binning、contigs binning和genes binning. Reads binning是依据reads的核酸序列组成和特点将所有reads分成若干个子集,然后进行后续宏基因组学分析.由于相关软件或者算法限制,reads binning的宏基因组数据利用率较低,故而并没有被广泛使用. Genes binning是将各个样本中的整体基因集,依据基因在各个样品中的丰度进行关联分析,利用相关性对基因进行聚类得到基因子集. Contigs binning是发展最快、应用最为广泛的序列分装手段. Contigs的序列长度远大于reads序列长度,依据核酸序列组成和特点的算法所得到的结果更加可靠且稳定;而对于数据的利用率,contigs binning也远大于reads binning.下面重点介绍contigs binning的方法和应用.
多种binning技术的整合有助于获得更多高质量的MAGs. Contig binning的方法主要分为3种:基于核酸组成(nucleotide composition(NC)-based)、基于丰度差异(differential abundance(DA)-based)和基于核酸组成及丰度(nucleotide composition and abundance(NCA)-based)[31]. NC法主要依赖寡核苷酸频率变化,DA法则依赖于微生物丰度不同的多个样本中contigs的覆盖度. NCA法结合了NC法和DA法,基于NC和DA创建复合距离矩阵进行后续聚类,是目前宏基因组binning的主流技术.基于NCA算法的软件工具有:MetaBAT[32]、CONCOCT[33]、GroopM[34]、MaxBin[35]和Databionuc ESOM工具[36]等. 2018年前后科研人员利用上述方法,大规模获取人体、肠道、土壤、海洋、污水处理反应池等生境中的微生物高质量MAGs[37-45].然而,横向比较发现针对不同生境的宏基因组数据,各种分装算法的表现并不相同,得到的MAGs在数量、污染度、基因组完整度指标上有明显的区别. 2018年5月,Sieber等开发出一种整合多种binning算法的DAS工具,通过与常见的5种单独binning算法进行比较,DAS获得了更多的高质量MAGs[41].同年9月,Uritskiy等也开发出整合多种binning算法的MetaWRAP工具[46],其在水体、土壤和肠道的测试宏基因组数据中表现明显优于单独的binning算法,相对于其他整合工具,如DAS和Binning_refiner[47],也略有优势.针对自然界中普遍存在水平基因转移现象[48],Song等开发出MetaCHIP工具,使用BLASTN软件鉴定MAGs中各个片段的物种来源,结合MAGs整体的物种注释信息,可以有效判别样品宏基因组中的水平基因转移特征[49].各类binning整合分析工具与基因元件鉴定工具的出现为揭示更多微生物未知信息提供了可能,同时也方便科研工作者整合结果,还原更加完整、真实的环境微生物菌群基因信息.
4 展望宏基因组学研究方法打破了基于微生物培养技术的传统微生物研究的困境,可以全面、真实地获取湖泊微生物多样性和功能多样性信息,同时也可以分析微生物与微生物之间、微生物与环境之间等的相互关系.利用高通量测序技术和生物信息学分析手段,湖泊微生物宏基因组学研究时间周期远小于传统微生物研究,一定程度上提高了研究效率.随着测序技术的不断发展,宏基因组学研究的成本在不断下降,微生物样本的宏基因组学研究将变得更加普及.与其他生境相比,湖泊微生物生态学研究处于落后状态,亟需开展大规模的微生物宏基因组学研究,通过广泛获得未知微生物高质量MAGs强化对湖泊微生物生态功能的认识.
微生物宏基因组学技术也存在方法自身的局限性[50](表 2).宏基因组结果可以表明功能基因存在与否,但无法确定功能基因的表达情况;测序结果容易受到污染序列的影响而降低研究的科学性和可靠性,避免或减少宏基因组样本中的污染或宿主序列仍旧是一个较大的难题;随着测序深度的不断增加,单个样品的宏基因组数据量可以达到几十甚至上百Gb,但是由于微生物培养技术的局限性和相关软件和数据库的限制,较大比例的物种信息和功能基因信息都无法获得注释,对测序数据的利用效率十分有限;利用宏基因组学研究湖泊微生物间相互关系时,采用数学统计和模型分析等手段常常会将样本微生物关系更复杂化.因此,通过微生物培养技术对微生物物种信息数据库和功能基因数据库进行扩充,对于宏基因组学研究是必需的.将宏基因组学研究与微生物培养技术、宏转录组学、宏蛋白组学、宏代谢组学等相结合,有望打破其当下的局限性,简化宏基因组学数据分析,提高研究结果的可靠性和科学性.随着生物信息学相关学科和技术的不断发展,宏基因组学技术将在湖泊微生物研究中发挥更为重要的作用,成为人类了解湖泊生态系统功能和维持机制的有力工具.
| 表 2 评估微生物群落的不同基因组分析方法优缺点 Tab. 2 Pros and cons of genomic analyses for evaluating microbial communities |
| [1] |
Wu QL, Xing P, Li HB et al. Impacts of regime shift between phytoplankton and macrophyte on the microbial community structure and its carbon cycling in lakes. Microbiology China, 2013, 40(1): 87-97. [吴庆龙, 邢鹏, 李化炳等. 草藻型稳态转换对湖泊微生物结构及其碳循环功能的影响. 微生物学通报, 2013, 40(1): 87-97.] |
| [2] |
Cui LJ. Evaluation on functions of Poyang Lake ecosystem. Chinese Journal of Ecology, 2004, 23(4): 47-51. [崔丽娟. 鄱阳湖湿地生态系统服务功能价值评估研究. 生态学杂志, 2004, 23(4): 47-51. DOI:10.3321/j.issn:1000-4890.2004.04.011] |
| [3] |
Azam F, Fenchel T, Field JG et al. The ecological role of water-column microbes in the sea. Mar Ecol Prog Ser, 1983, 10: 257-263. DOI:10.3354/meps010257 |
| [4] |
Wu QL, Jiang HL. Microbiological research on Chinese lakes. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 273-279. [吴庆龙, 江和龙. 中国湖泊微生物组研究. 中国科学院院刊, 2017, 32(3): 273-279.] |
| [5] |
Riesenfeld CS, Schloss PD, Handelsman J. Metagenomics:genomic analysis of microbial communities. Annu Rev Genet, 2004, 38: 525-552. DOI:10.1146/annurev.genet.38.072902.091216 |
| [6] |
Prakash O, Shouche Y, Jangid K et al. Microbial cultivation and the role of microbial resource centers in the omics era. Appl Microbiol Biotechnol, 2013, 97(1): 51-62. DOI:10.1007/s00253-012-4533-y |
| [7] |
Sanz JL, Köchling T. Molecular biology techniques used in wastewater treatment:An overview. Process Biochemistry, 2007, 42(2): 119-133. DOI:10.1016/j.procbio.2006.10.003 |
| [8] |
Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiol Rev, 1995, 59(1): 143-169. |
| [9] |
Handelsman J. Metagenomics:application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev, 2004, 68(4): 669-685. DOI:10.1128/MMBR.69.1.195.2005 |
| [10] |
Zengler K, Palsson BO. A road map for the development of community systems (CoSy) biology. Nature Reviews Microbiology, 2012, 10(5): 366. DOI:10.1038/nrmicro2763 |
| [11] |
Sun X, Gao Y, Yang YF. Recent advancement in microbial environmental research using metagenomics tools. Biodiversity Science, 2013, 21(4): 393-400. [孙欣, 高莹, 杨云锋. 环境微生物的宏基因组学研究新进展. 生物多样性, 2013, 21(4): 393-400.] |
| [12] |
De Corte D, Srivastava A, Koski M et al. Metagenomic insights into zooplankton-associated bacterial communities. Environmental Microbiology, 2018, 20(2): 492-505. DOI:10.1111/1462-2920.13944 |
| [13] |
Hess M, Sczyrba A, Egan R et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen. Science, 2011, 331(6016): 463-467. DOI:10.1126/science.1200387 |
| [14] |
Bowers RM, Kyrpides NC, Stepanauskas R et al. Minimum information about a single amplified genome (MISAG) and a metagenome-assembled genome (MIMAG) of bacteria and archaea. Nature Biotechnology, 2017, 35(8): 725. DOI:10.1038/nbt.3893 |
| [15] |
Parks DH, Imelfort M, Skennerton CT et al. CheckM:assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [16] |
Vavourakis CD, Andrei AS, Mehrshad M et al. A metagenomics roadmap to the uncultured genome diversity in hypersaline soda lake sediments. Microbiome, 2018, 6(1): 168. DOI:10.1186/s40168-018-0548-7 |
| [17] |
Arora-Williams K, Olesen SW, Scandella BP et al. Dynamics of microbial populations mediating biogeochemical cycling in a freshwater lake. Microbiome, 2018, 6(1): 165. DOI:10.1186/s40168-018-0556-7 |
| [18] |
Cabello-Yeves PJ, Zemskaya TI, Rosselli R et al. Genomes of novel microbial lineages assembled from the sub-ice waters of Lake Baikal. Appl Environ Microbiol, 2018, 84(1): e02132-17. DOI:10.1128/AEM.02132-17 |
| [19] |
Yau S, Lauro FM, Williams TJ et al. Metagenomic insights into strategies of carbon conservation and unusual sulfur biogeochemistry in a hypersaline Antarctic lake. The ISME Journal, 2013, 7(10): 1944-1961. DOI:10.1038/ismej.2013.69 |
| [20] |
Lauro FM, Demaere MZ, Yau S et al. An integrative study of a meromictic lake ecosystem in Antarctica. The ISME Journal, 2010, 5(5): 879-895. DOI:10.1038/ismej.2010.185 |
| [21] |
Alvarenga DO, Fiore FM, Varani AM. A Metagenomic approach to cyanobacterial genomics. Front Microbiol, 2017, 8: 809. DOI:10.3389/fmicb.2017.00809 |
| [22] |
Xie M, Ren M, Yang C et al. Metagenomic analysis reveals symbiotic relationship among bacteria in Microcystis-dominated community. Front Microbiol, 2016, 7: 56. DOI:10.3389/fmicb.2016.00056 |
| [23] |
Zhang JY. Metagenomic studies on cyanobacterial blooms in Lake Taihu[Dissertation]. Nanjing: South-East University, 2018. [张军毅.太湖蓝藻水华的宏基因组学研究[学位论文].南京: 东南大学, 2018. ]
|
| [24] |
He S, Stevens SLR, Chan LK et al. Ecophysiology of freshwater Verrucomicrobia inferred from metagenome-assembled genomes. mSphere, 2017, 2(5): e00277-17. DOI:10.1128/mSphere.00277-17 |
| [25] |
Cuadrat RRC, Ionescu D, Dávila AMR et al. Recovering genomics clusters of secondary metabolites from lakes using genome-resolved metagenomics. Frontiers in Microbiology, 2018, 9: 251. DOI:10.3389/fmicb.2018.00251 |
| [26] |
Mehrshad M, Salcher MM, Okazaki Y et al. Hidden in plain sight-highly abundant and diverse planktonic freshwater Chloroflexi. Microbiome, 2018, 6(1): 176. DOI:10.1186/s40168-018-0563-8 |
| [27] |
Andrei A Ç, Salcher MM, Mehrshad M et al. Niche-directed evolution modulates genome architecture in freshwater Planctomycetes. The ISME Journal, 2019, 13(4): 1056-1071. DOI:10.1038/s41396-018-0332-5 |
| [28] |
Pop M, Salzberg SL. Bioinformatics challenges of new sequencing technology. Trends in Genetics, 2008, 24(3): 142-149. DOI:10.1016/j.tig.2007.12.006 |
| [29] |
Rhoads A, Au KF. PacBio sequencing and its applications. Genomics, Proteomics & Bioinformatics, 2015, 13(5): 278-289. DOI:10.1016/j.gpb.2015.08.002 |
| [30] |
Clarke J, Wu HC, Jayasinghe L et al. Continuous base identification for single-molecule nanopore DNA sequencing. Nature Nanotechnology, 2009, 4(4): 265-270. DOI:10.1038/nnano.2009.12 |
| [31] |
Sangwan N, Xia F, Gilbert JA. Recovering complete and draft population genomes from metagenome datasets. Microbiome, 2016, 4(1): 8. DOI:10.1186/s40168-016-0154-5 |
| [32] |
Kang DD, Froula J, Egan R et al. MetaBAT, an efficient tool for accurately reconstructing single genomes from complex microbial communities. Peer J, 2015, 3: e1165. DOI:10.7717/peerj.1165 |
| [33] |
Alneberg J, Bjarnason BS, de Bruijn I et al. CONCOCT:clustering contigs on coverage and composition. Quantitative Biology, 2014, 11(11): 1144-1146. |
| [34] |
Imelfort M, Parks D, Woodcroft BJ et al. GroopM:an automated tool for the recovery of population genomes from related metagenomes. Peer J, 2014, 2: e603. DOI:10.7717/peerj.603 |
| [35] |
Wu YW, Simmons BA, Singer SW. MaxBin 2.0:an automated binning algorithm to recover genomes from multiple metagenomic datasets. Bioinformatics, 2015, 32(4): 605-607. DOI:10.1093/bioinformatics/btv638 |
| [36] |
Ultsch A, Mörchen F. ESOM-Maps:tools for clustering, visualization, and classification with emergent SOM. Germany:Data Bionics Research Group, University of Marburg, 2005. |
| [37] |
Parks DH, Rinke C, Chuvochina M et al. Recovery of nearly 8, 000 metagenome-assembled genomes substantially expands the tree of life. Nature Microbiology, 2017, 2(11): 1533-1542. DOI:10.1038/s41564-017-0012-7 |
| [38] |
Pasolli E, Asnicar F, Manara S et al. Extensive unexplored human microbiome diversity revealed by over 150, 000 genomes from metagenomes spanning age, geography, and lifestyle. Cell, 2019, 176(3): 649-662. e20. DOI:10.1016/j.cell.2019.01.001 |
| [39] |
Almeida A, Mitchell AL, Boland M et al. A new genomic blueprint of the human gut microbiota. Nature, 2019. DOI:10.1038/s41586-019-0965-1 |
| [40] |
Stewart RD, Auffret MD, Warr A et al. Assembly of 913 microbial genomes from metagenomic sequencing of the cow rumen. Nature Communications, 2018, 9(1): 870. DOI:10.1038/s41467-018-03317-6 |
| [41] |
Sieber CMK, Probst AJ, Sharrar A et al. Recovery of genomes from metagenomes via a dereplication, aggregation and scoring strategy. Nature Microbiology, 2018, 3(7): 836-843. DOI:10.1038/s41564-018-0171-1 |
| [42] |
Zhang Y, Hua Z, Lu H et al. Elucidating functional microorganisms and metabolic mechanisms in a novel engineered ecosystem integrating C, N, P and S biotransformation by metagenomics. Water Res, 2019, 148: 219-230. DOI:10.1016/j.watres.2018.10.061 |
| [43] |
Campanaro S, Treu L, Kougias PG et al. Metagenomic binning reveals the functional roles of core abundant microorganisms in twelve full-scale biogas plants. Water Res, 2018, 140: 123-134. DOI:10.1016/j.watres.2018.04.043 |
| [44] |
Tully BJ, Graham ED, Heidelberg JF. The reconstruction of 2, 631 draft metagenome-assembled genomes from the global oceans. Scientific Data, 2018, 5: 170203. DOI:10.1038/sdata.2017.203 |
| [45] |
DeMaere MZ, Darling AE. bin3C:exploiting Hi-C sequencing data to accurately resolve metagenome-assembled genomes. Genome Biology, 2019, 20(1): 46. DOI:10.1186/s13059-019-1643-1 |
| [46] |
Uritskiy GV, DiRuggiero J, Taylor J. MetaWRAP-a flexible pipeline for genome-resolved metagenomic data analysis. Microbiome, 2018, 6: 158. DOI:10.1186/s40168-018-0541-1 |
| [47] |
Song WZ, Thomas T. Binning_refiner:improving genome bins through the combination of different binning programs. Bioinformatics, 2017, 33(12): 1873-1875. DOI:10.1093/bioinformatics/btx086 |
| [48] |
Dagan T, Artzy-Randrup Y, Martin W. Modular networks and cumulative impact of lateral transfer in prokaryote genome evolution. Proceedings of the National Academy of Sciences, 2008, 105: 10039-10044. DOI:10.1073/pnas.0800679105 |
| [49] |
Song W, Wemheuer B, Zhang S et al. MetaCHIP:community-level horizontal gene transfer identification through the combination of best-match and phylogenetic approaches. Microbiome, 2019, 7: 36. DOI:10.1186/s40168-019-0649-y |
| [50] |
Knight R, Vrbanac A, Taylor BC et al. Best practices for analysing microbiomes. Nature Reviews Microbiology, 2018, 17: 410-422. DOI:10.1038/s41579-018-0029-9 |
 2020, Vol. 32
2020, Vol. 32 

