(2: 中国科学院大学,北京 100049)
(3: 河海大学水文水资源学院,南京 210098)
(2: University of Chinese Academy of Sciences, Beijing 100049, P.R.China)
(3: College of Hydrology and Water Resources, Hohai University, Nanjing 210098, P.R.China)
沉水植物是水生态系统中重要的初级生产者,具有净化水体、降低营养盐浓度等重要生态功能[1]. 叶片是沉水植物重要的器官,可以进行光合作用,调节植物自身的资源和能量[2];同时它们能提供一个较大面积的水-植物界面[3],并在界面上形成生物膜,为细菌附着定殖提供营养物质和微生态位,形成叶片附着细菌群落[4]. 叶片附着细菌数量众多、种类丰富[3],这些细菌能为沉水植物提供营养物质,部分细菌还能帮助植物抵抗病原体,对植物生长和健康十分重要[5]. 同时,叶片附着细菌作为湖泊生态系统中的重要组成部分,在促进水体中有机物降解以及营养物质循环过程中都发挥着重要作用[6]. 因此研究叶片附着细菌群落结构及多样性,可以丰富人们对这种特殊生境下生物多样性的认识,对于保护水生植物健康、维护湖泊生态环境有深远意义[7].
有研究发现,叶片附着细菌群落和浮游细菌群落之间存在一些共有微生物[8]. 生活在周围水域的浮游细菌一般被认为是沉水植物叶片附着细菌的主要种子库,对叶片附着细菌群落组成有重要影响[9],沉水植物可以通过不同叶片上复杂的物理或生化特性来选择宿主特异性的细菌群落[10]. 同样,浮游细菌群落也会受到沉水植物的影响[11],叶片附着细菌可能扩散到浮游细菌群落中. 同时,叶片附着细菌和周围水体中浮游细菌群落也存在一些截然不同的细菌类群[12-13],这表明细菌在两种生境之间的扩散或交换可能是有限的,物理屏障或宿主选择性差异使得叶片附着细菌群落和浮游细菌群落之间产生分化[10]. 然而,目前的研究缺乏对这两个相互影响的细菌群落结构和功能之间异同的理解.
影响植物叶片附着细菌群落的因素,过去通常用环境因素来解释[14],最近越来越多的研究发现宿主植物自身的生化特性对叶片附着细菌群落有重要影响. 刘凯辉等[15]观测金鱼藻(Ceratophyllum demersum)、伊乐藻(Elodea nuttallii)和菹草(Potamogeton crispus)3种沉水植物叶片附着细菌的分布特征,发现不同植物种类通过叶表物理形态和结构差异显著影响叶片附着细菌群落. Cai等[16]对太湖不同地点的马来眼子菜(Potamogeton malaianus)叶片附着细菌群落结构特征进行研究,发现不同取样点马来眼子菜附着细菌组成具有高度的相似性,推断其也具有一定的宿主选择作用. 但这些研究大多集中于对沉水植物叶表附着细菌种类、密度、生物量等指标的比较,目前对于不同植物宿主如何影响叶片附着细菌群落组成和多样性以及细菌群落物种相关性网络的研究仍然十分匮乏.
本研究选取苦草(Vallisneria natans)、黑藻(Hydrilla verticillata)、穗状狐尾藻(Myriophyllum spicatum)等典型沉水植物,在东太湖试验站构建中尺度模拟实验体系,分别设置单物种体系(低植物物种多样性)和多物种体系(高植物物种多样性),模拟不同植物多样性配置和植物种类对水体浮游细菌和叶片附着细菌群落的影响,应用高通量测序解析浮游细菌群落和植物叶片附着细菌群落差异. 本研究旨在回答以下科学问题:(1)不同植物物种多样性体系中,浮游细菌和植物叶片附着细菌群落的差异如何?(2)在高植物物种多样性体系中,不同种类植物叶片附着细菌群落的差异如何?(3)不同植物叶片附着细菌物种共现网络中是否存在关键物种,关键物种是否不同?
1 材料与方法 1.1 试验设计本研究所用苦草、黑藻、穗状狐尾藻和微齿眼子菜(Potamogeton maackianus)均采集于中国科学院太湖湖泊生态系统研究站(31°2′0.75″N, 120°25′17.23″E)的不同围隔体系. 实验设置8个围隔,分别为4个低植物多样性处理和4个高植物物种多样性处理. 围隔长2 m、宽2 m,深1.6 m左右. 围隔采用防水PVC材料,底部插入沉积物中,上部高出水面. 围隔中混匀的沉积物理化性状基本一致;实验用水来自与东太湖连通的池塘,经孔径1 mm的筛网过滤后用水泵打入围隔中. 体系中植物由人工均匀种植,初始种植植物株数相同. 低植物物种多样性处理只种植1种沉水植物苦草,高植物物种多样性处理种植苦草、穗状狐尾藻、微齿眼子菜、黑藻、金鱼藻、轮藻(Charophyceae)、水盾草(Cabomba)和篦齿眼子菜(Potamogeton pectinatus)8种沉水植物.
1.2 样品采集2021年1月24日,于观测站进行样品采集. 使用采水器采集每个围隔体系内的水下约0.5~0.6 m处水样,用灭菌的聚乙烯瓶迅速收集500 mL水样. 使用采草器在距水面0.6~0.9 m处采集每种植物的叶片,取中间绿色长势良好的部位进行采集. 在低植物多样性处理围隔体系内采集苦草叶片样品,在高植物物种多样性处理围隔体系中采集苦草、黑藻、穗状狐尾藻和微齿眼子菜叶片样品. 每种植物采集6片叶片合并作为一个样品,用无菌水冲洗3次后立即置于50 mL离心管中. 将采集的样品放置于4℃保温箱内冷藏,当天运回实验室以备后续处理.
1.3 样品预处理及理化指标测定将植物叶片放在含有35 mL PBS缓冲液(130 mmol/L NaCl,7 mmol/L Na2HPO4,3 mmol/L NaH2PO4,pH=7.0,0.02 % Silwet L-77)的50 mL离心管中,漩涡振荡5 min;用灭菌的镊子将叶片移入另一个干净的含35 mL PBS缓冲液的50 mL离心管中,漩涡振荡5 min. 将前两步洗脱的PBS缓冲液通过0.2 μm的滤膜过滤(milipore,USA)[9],所得滤膜用来提取DNA.
使用YSI 6600型水质分析仪现场测定水温(WT)、溶解氧(DO)、pH值等指标[17];用乙醇萃取分光光度法测定水体中叶绿素a(Chl.a)浓度,分别采用碱性过硫酸钾消解紫外分光光度法(HJ 636-2012)和钼酸铵分光光度法(GB 11893-1989)测定总氮(TN)和总磷(TP)浓度;用MultiN/C2100TOC/TN测定溶解性有机碳(DOC)浓度,用流动注射仪(San++,SKALAR,Netherlands)测定氨氮(NH3-N)、硝态氮(NO3--N)、亚硝态氮(NO2--N)浓度[18].
1.4 DNA提取和高通量测序使用E.Z.N.A.Ⓡ Water DNA试剂盒(Omega,USA)提取植物叶片附着细菌和浮游细菌的DNA. 用NanoDrop ND2000(Thermo Fisher Scientific,Wilmington,DE,USA)分光光度计测定其浓度和纯度,并通过0.8 % (W/V)琼脂糖凝胶电泳进行观察[19].
采用正向引物515F(5′-GTGYCAGCMGCCGCGGTAA-3′)和反向引物806R(5′-GGACTACNVGGGTWTCTAAT-3′)对细菌16S rRNA基因的V4区进行PCR扩增. PCR扩增体系(20 μL:0.4 μL FastPfu聚合酶、0.8 μL正向引物(5 μmol/L)、0.8 μL反向引物(5 μmol/L)、4 μL 5×Fast Pfu缓冲液、2 μL dNTPs(2.5 mmol/L)和10 ng模板DNA). 反应条件为95℃ 2 min;95℃ 30 s;55℃ 30 s;72℃ 30 s,共25个循环;72℃ 5 min,10℃终止反应[20]. 用2 % 琼脂糖凝胶回收PCR产物,并用Qiagen凝胶提取试剂盒(Qiagen,德国)进行纯化. 利用QubitⓇ3.0(Life Invitrogen)对纯化的PCR产物进行定量,利用Illumina公司的MiSeq-PE300平台进行测序,由广东美格基因科技有限公司完成.
使用Trimmomatic(v.0.33)[21]对原始测序序列进行质控. 使用FLASH(v.1.2.11)[22]软件进行拼接. 使用USEARCH软件(v.4.2.52)[23]根据97 % 的相似度对序列进行OTU(operational taxonomic unit)聚类,并在聚类的过程中使用带有denovo策略的UCHIME算法[24]去除单一序列和嵌合体. 应用核糖体数据库项目(RDP)分类器基于SILVA数据库(1.32版本)检索每个OTU的分类信息[25],以80 % 的阈值确定OTU的分类学属性[26].
1.5 网络分析在本研究中,基于Sparse correlations for compositional data(SparCC)(https://bitbucket.org/yonatanf/sparcc)方法分析OTU两两之间的关系,选择相对丰度> 0.02 % 以及在1/2以上样本中存在的OTU进行网络分析,选取标准为强相关性(SparCC|r|> 0.60)和统计显著性(P<0.01)[27-28]. 同时将环境变量整合到网络分析中,以揭示节点和环境变量之间的关系. 通过Cytoscape (v3.6.1)进行网络分析结果的可视化. 计算Zi(模块内连通性)和Pi(模块间连通性),以显示网络节点的拓扑作用. 区分网络中节点的4个子类别的标准:外围节点(Zi ≤ 2.5,Pi ≤ 0.62),连接点(Zi ≤ 2.5,Pi ≥ 0.62),模块核心(Zi ≥ 2.5,Pi ≤ 0.62)和网络核心(Zi ≥ 2.5,Pi ≥ 0.62).
基于高植物物种多样性体系中4种植物,分别与水体构建了4个共现网络. 使用R(v3.6.1)中的“igraph”包(v1.2.4.1)计算模块化指数、平均度等网络拓扑参数. 模块化网络通常由不同的模块组成,网络中的模块是一组互相连接度高的节点,它们与其它模块中的节点连接较少;网络模块化指数旨在衡量网络划分为模块的程度[29-30]. 模块化指数>0.4表示网络达到了一定的模块化程度,具有模块性. 平均度也称为平均连通性,描述网络中每个节点的平均连接边数,可以指示网络复杂度[31]. 此外,使用R中的“igraph”包[32]生成了1000个随机Erdös-Rényi网络,其节点和边数与观察网络相同,并计算它们的拓扑参数. 在R软件中使用Z-test测试观察网络和随机网络之间拓扑特征的差异[33].
1.6 统计学分析测序结果通过生物信息学软件QIIME、Mothur及统计软件R等处理分析[34-35]. 使用QIIME脚本“alpha_diversity.py”命令[36]计算OTU丰富度、系统发育多样性. 使用QIIME脚本“beta_diversity.py”命令[36]进行β多样性分析. 水体理化指标、α多样性、β多样性箱型图和分类学信息图绘图使用GraphPad Prism 8完成. 同时在SPSS(v.20.0)中使用独立样本t-检验和单因素方差分析检验每组数据之间差异的显著性. 使用非度量多维尺度分析(NMDS)对不同样品间细菌群落结构可视化,并使用R软件的“vegan”包[36]进行相似性分析(ANOSIM),对细菌群落结构在不同生境(植物、水体)间的差异进行显著性检验.
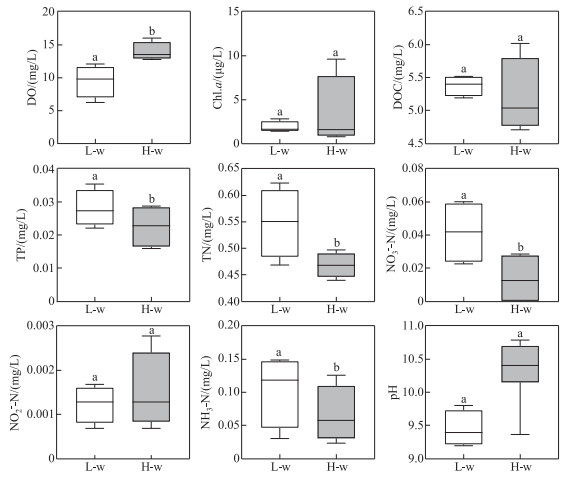
2 结果 2.1 水体理化指标不同围隔体系的水体理化特征如图 1所示,理化指标在不同的植物物种多样性体系中均存在差异. 单因素ANOVA检验的结果表明,DO、TP、TN、NO3--N、NH3-N浓度在不同体系中均存在显著性差异(P < 0.05),其中TP、TN、NO3--N、NH3-N浓度从低植物物种多样性体系到高植物物种多样性体系呈下降趋势,相反,DO呈上升趋势.
|
图 1 植物种植体系水体理化因子(每种处理设置4个重复. L-w: 低植物物种多样性围隔水体;H-w: 高植物物种多样性围隔水体. 箱型图上不同小写字母表示不同物种多样性体系水体环境因子之间差异显著. 独立样本t-检验,P < 0.05) Fig.1 Physical and chemical factors of water in different plant diversity systems (4 replicates for each water sample. L-w: water with low submerged macrophytes diversity; H-w: water with high submerged macrophytes diversity. Different letters above columns indicate significant difference between different submerged macrophytes diversity systems. Independent sample t-test, P < 0.05) |
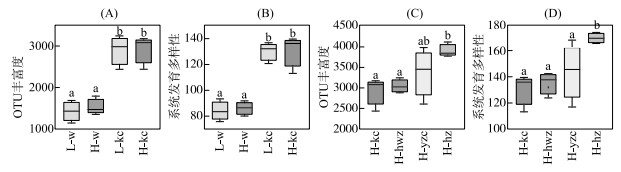
从8个围隔的28个叶片样品中共获得了7903583条有效16S rRNA基因序列和11168个OTU. α多样性分析显示,浮游细菌群落OTU丰度和系统发育指数多样性显著低于植物叶片附着细菌群落(P < 0.05)(图 2). 高植物多样性体系中的浮游细菌群落和植物叶片附着细菌群落α多样性稍高于低植物多样性体系但不显著(P > 0.05)(图 2A, B). 高植物多样性体系中黑藻叶片附着细菌群落α多样性显著高于其它植物(P < 0.05),苦草和穗状狐尾藻叶片附着细菌群落α多样性较低(图 2C, D).
|
图 2 不同植物物种多样性体系中浮游细菌和植物叶片附着细菌群落α多样性(每组样品有4个重复. L: 低植物物种多样性;H: 高植物物种多样性;w: 水体;kc: 苦草;hwz: 穗状狐尾藻;yzc: 微齿眼子菜;hz: 黑藻. 箱型图上不同的小写字母表示显著差异. 单因素ANOVA分析,P < 0.05) Fig.2 The α diversity of bacterioplankton and epiphytic bacterial communities in different systems (There are 4 replicates for each water sample. L-w: water with low submerged macrophytes diversity; H-w: water with high submerged macrophytes diversity; w: water; kc: Vallisneria natans; hwz: Myriophyllum spicatum; yzc: Potamogeton maackianus and hz: Hydrilla verticillata. Different letters above columns indicate significant difference. One-way ANOVA analysis, P < 0.05) |
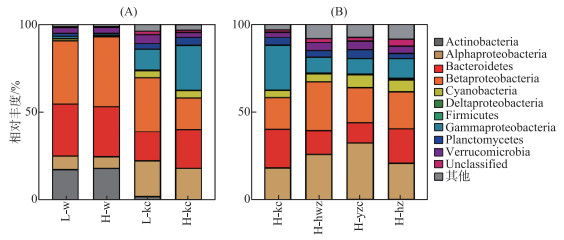
在门分类水平上,浮游细菌群落和苦草叶片附着细菌群落的物种组成不同(图 3A),不同种类沉水植物叶片附着细菌群落组成也存在一定差异(图 3B). α-变形菌纲(α-proteobacteria,20.35 %)、β-变形菌纲(β-proteobacteria,30.59 %)、γ-变形菌纲(γ-proteobacteria,11.91 %)和拟杆菌门(16.51 %)是苦草叶片附着细菌群落的优势菌门(纲). 相比之下,浮游细菌群落的优势菌门(纲)是拟杆菌门(29.55 %)和β变形菌纲(35.86 %)(图 3). 细菌群落相对丰度top5 OTU表显示,植物叶片附着细菌群落相对丰度top5 OTU有3个是α-变形菌纲,而浮游细菌群落相对丰度的top5 OTU有5个属于拟杆菌. 其中Rhodobacteraceae(红杆菌科,隶属于α-变形菌纲)是叶片附着细菌top5 OTU独有(表 1),Actinomycetales(放线菌目,隶属于放线菌科)和Polynucleobacter(多核杆菌属,隶属于β-变形菌纲)是浮游细菌top5 OTU独有(表 1). 还有一些细菌如Flavobacterium(黄杆菌属,隶属于拟杆菌门)在浮游细菌群落和植物叶片附着细菌群落中都有分布(表 1).
|
图 3 不同样品组在门分类水平上的细菌群落组成(L: 低植物物种多样性;H: 高植物物种多样性;w: 水体;kc: 苦草;hwz: 穗状狐尾藻;yzc: 微齿眼子菜;hz: 黑藻) Fig.3 Bacterial community composition of different sample groups at the phylum level (class for Proteobacteria) (Relative abundance of dominant bacteria phyla in bacterioplankton and epiphytic bacterial communities. L-w: water with low submerged macrophytes diversity; H-w: water with high submerged macrophytes diversity; w: water; kc: Vallisneria natans; hwz: Myriophyllum spicatum; yzc: Potamogeton maackianus and hz: Hydrilla verticillata) |
| 表 1 不同植物物种多样性体系中浮游细菌和植物叶片附着细菌群落相对丰度前5的OTUs Tab. 1 The top5 OTUs in bacterioplankton and epiphytic bacteria in different plant diversity systems arranged by their relative abundance |
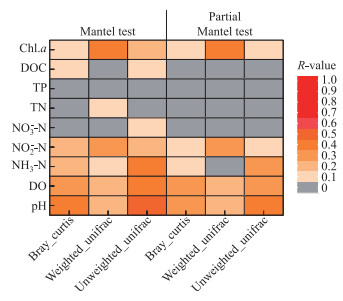
基于未加权UniFrac距离的NMDS分析表明,浮游细菌群落和苦草叶片附着细菌群落沿着NMDS1轴区分明显(图 4). ANOSIM分析结果显示(表 2),浮游细菌和植物叶片附着细菌群落结构存在显著差异(P < 0.05). 高植物物种多样性体系中,除了微齿眼子菜和穗状狐尾藻之外,不同植物叶片附着细菌群落结构均存在显著差异(P < 0.05). 箱型图显示不同体系的浮游细菌群落和苦草叶片附着细菌群落β多样性之间无显著差异. 高植物多样性体系中不同植物叶片附着细菌群落之间,微齿眼子菜的叶片附着细菌群落β多样性显著最高,黑藻显著最低(P < 0.05)(图 4). 环境因子影响叶片附着细菌群落β多样性,其中NH3-N、NO2--N、Chl.a与植物叶片附着细菌群落有显著关联(图 5).

|
图 4 不同植物物种多样性体系中浮游细菌和植物叶片附着细菌群落β多样性(L: 低植物物种多样性;H: 高植物物种多样性;w: 水体;kc: 苦草;hwz: 穗状狐尾藻;yzc: 微齿眼子菜;hz: 黑藻. 箱型图上不同的小写字母表示显著差异. 单因素ANOVA分析,P < 0.05) Fig.4 The β diversity of bacterioplankton and epiphytic bacterial communities in different plant diversity systems (L-w: water with low submerged macrophytes diversity; H-w: water with high submerged macrophytes diversity; w: water; kc: Vallisneria natans; hwz: Myriophyllum spicatum; yzc: Potamogeton maackianus and hz: Hydrilla verticillata. Different letters above columns indicate significant difference. One-way ANOVA analysis, P < 0.05) |
| 表 2 相似性分析(ANOSIM)测试不同植物物种多样性体系中浮游、植物叶片附着细菌群落组成的差异 Tab. 2 Analysis of similarities to test the differences in bacterioplankton and epiphytic bacterial community composition in different systems |
|
图 5 Mantel和偏Mantel检验分析叶片附着细菌群落结构与环境因子之间的关系(显著相关(P < 0.05)用橙黄色表示,不显著相关用灰色方块表示) Fig.5 Mantel and partial Mantel tests for the relationship between dissimilarities of epiphytic bacteria and environmental variables(Significant correlations (P < 0.05) are shown with yellow-orange colors, and insignificant results are indicated by gray squares) |
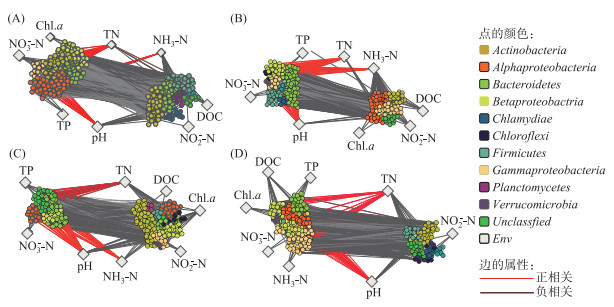
基于高植物物种多样性体系中不同植物(苦草、黑藻、微齿眼子菜和穗状狐尾藻)构建4个不同的细菌物种共现网络,为了探索环境因素对植物叶片细菌的影响,将环境变量加入到网络分析中. 将OTU节点按照门水平分类赋予不同的颜色. 结果显示,总氮浓度与苦草(86.36 %)、狐尾藻(82.35 %)、微齿眼子菜(56 %)叶片细菌群落的相关关系以正相关关系为主(图 6A,B,C),与黑藻(40.82 %)叶片细菌网络以负相关关系为主(图 6D). 氨氮浓度与微齿眼子菜(75 %)叶片细菌群落的相关关系以正相关关系为主(图 6C). 观察到4个网络的拓扑特征与相应的随机网络显著不同(P < 0.05),表明细菌群落的共现网络是非随机的(表 3). 所有植物的叶片附着细菌群落网络正相关关系占比较高,平均度较高. 不同植物网络显示,苦草(58.79 %)和黑藻(65.47 %)网络中的相关关系以正相关为主,苦草模块化指数更高,穗状狐尾藻平均度较高(表 3).
|
图 6 基于SparCC相关分析的环境因子和细菌物种-物种网络特征((A)苦草;(B)穗状狐尾藻;(C)黑藻;(D)微齿眼子菜. 连接两个节点的每条边代表正(红线)或负(黑线)相关关系) Fig.6 Characteristics of environmental factors and bacterial species-species networks based on SparCC correlation analysis((A) Vallisneria natans; (B) Myriophyllum spicatum; (C) Hydrilla verticillata; (D) Potamogeton maackianus. The red solid lines indicate positive correlations and the black lines indicate negative correlations) |
| 表 3 高植物物种多样性体系植物叶片附着细菌群落观察网络及其相关随机网络的拓扑参数 Tab. 3 Topological parameters of epiphytic bacterial communities' observation network and random network |
高植物物种多样性体系中不同植物网络的关键物种信息表显示(表 4),在苦草网络中的7个关键OTU中,有4个来自拟杆菌门. 在穗状狐尾藻叶片附着细菌网络中关键OTU中,OTU112295来自疣微菌门(Verrucomicrobiae)和OTU102189来自蓝细菌门(Cyanobacteria),它们属于相对丰度top5 OTU优势细菌(表 1). 在黑藻叶片附着细菌网络中的7个关键OTU中,4个来自β-变形菌纲,另有OTU270来自浮霉菌门(Planctomycetes),属于相对丰度top5 OTU优势细菌(表 1). 在微齿眼子菜叶片附着细菌网络中的9个关键OTU中,4个来自α-变形菌纲;其中OTU93909和OTU158474属于相对丰度top5 OTU优势细菌(表 1). 在所有植物叶片附着细菌的综合网络中,15个关键OTU有10个来自α-变形菌纲.
| 表 4 高植物物种多样性体系中不同植物物种共现网络中的模块核心和连接点* Tab. 4 Module hubs and connectors in the species-species networks in high submerged macrophytes diversity system |
研究结果显示,种植更多种类的沉水植物能显著降低水体中TN、TP浓度(图 1). 与浮游细菌群落相比,叶片附着细菌群落表现出较高的α多样性(OTU丰富度和系统发育多样性)(图 2). 叶片附着细菌在水体和植物组织之间的界面生活方式赋予了其从这2种来源获取营养的竞争优势,宿主植物可通过分泌物或光合产物提供养分来支持更高多样性的细菌群落[37],同时为细菌提供更多的生存空间[10]. 系统发育多样性与群落内物种之间的关键功能差异相关,可能代表植物叶片附着细菌和浮游细菌功能上的差异[38]. 此外,叶片的内生细菌向外迁移到叶表面后也可能对附着细菌群落做出特殊贡献[39],这可以部分增加附着细菌群落丰富度,并形成不同的群落组成. 同时浮游植物群落可以显著影响浮游细菌的多样性[37],沉水植物因自身竞争和化感作用可能抑制浮游植物的生长,进而影响浮游细菌多样性. 在不同植物物种多样性体系的对比中,观察到高植物物种多样性体系中植物叶片附着细菌群落的α多样性较高. 沉水植物叶面可能有大量的碳源供叶片附着细菌利用[40],与单种植物相比,更多种类的沉水植物能提供更多种类的营养物质[41-42],来支持更多的细菌物种存活. 其次,采样的季节(冬天)水生植物大都处在衰亡初期,相比其它生长阶段,衰亡初期的植物能够为附着在其表面的细菌提供更多的无机和有机营养物质[43],随着植物种类增加,高植物物种多样性体系有更多样的植物处于衰亡期,从而能提供更多营养物质,使得体系内植物叶片附着细菌群落有更高的α多样性指数[44].
NMDS分析结果表明,浮游细菌群落与植物叶片附着细菌群落结构之间的相似度较低(图 4),ANOSIM分析结果(表 2)验证了它们之间均存在显著差异. 一方面,扩散限制可能导致浮游细菌群落和植物叶片附着细菌群落产生差异. 和根际土壤-植物连续体类似[45],水生植物叶际分泌物、营养成分、细菌等会在水体中进行迁移扩散,可能会在植物叶片-水体连续体中形成空间梯度. 另一方面,通过细菌群落溯源分析,发现水环境可能是叶面附着细菌的主要来源,平均贡献65.79 % 的附着细菌群落[46]. 细菌在水体和叶片表面生物膜这两种生境之间的扩散或交换也是有限的,宿主选择性的差异使得叶片附着细菌群落和浮游细菌群落之间产生分化[10],植物宿主选择效应是塑造沉水植物叶片附着细菌群落的另一个重要因子.
在门分类水平上,相比植物叶片附着细菌群落,浮游细菌群落的拟杆菌门和β-变形菌纲相对丰度较高(图 3). 拟杆菌门在以高等水生植物为初级生产者的清水态湖泊中居多[47],可以协助部分有机大分子物质分解. 而变形菌门有显著的固氮作用,广泛分布于全球不同类型的湖泊中,数量极为丰富[10]. 随着植物物种多样性增加,浮游细菌的拟杆菌门比例降低. 李琳琳等[48]的研究发现营养盐浓度影响浮游细菌的群落组成,拟杆菌门数量随着营养盐浓度减弱而减少. 植物叶片附着细菌群落相对丰度top5 OTU中有3个属于α-变形菌纲,其中红杆菌(隶属α-变形菌纲)是叶片附着细菌top5 OTU独有的(表 1),它在附着细菌生物膜早期形成过程中发挥着关键作用[49]. 而在浮游细菌群落中序列数相对丰度top5 OTU有3个属于β-变形菌纲,它们当中比如放线菌和多核杆菌(隶属β-变形菌纲)是浮游细菌top5 OTU独有的(表 1). 这些细菌属于广泛分布的淡水湖泊浮游细菌典型种群,浮游细菌群落独有OTU中也表明这些细菌或许不具备在叶片表面成为优势物种的能力. 另外,还有一些细菌,如黄杆菌(隶属拟杆菌)(表 1),在浮游细菌群落和植物叶片附着细菌群落中都有分布,它们可能具备多样的代谢方式和更强的生存适应能力.
3.2 不同水生植物叶片附着细菌群落存在差异高植物物种多样性体系中,黑藻叶片附着细菌群落α多样性显著高于其它植物(P < 0.05). 有研究发现,沉水植物与叶片附着细菌发生相互作用时能够分泌化感物质,这些化感物质能够对细菌产生影响[50]. 对于化感活性强的植物,大量胞外分泌物可能有针对性地抑制一种或者几种细菌的生长,降低细菌群落的多样性[51]. 本研究中的4种沉水植物,黑藻的化感活性较弱,微齿眼子菜和穗状狐尾藻的化感活性较强[52],这有可能导致黑藻叶片附着细菌多样性明显高于另外几种沉水植物. 尽管本研究没有分析叶片附着真菌的组成,但有研究证明黑藻叶片分泌物促进了细菌-真菌的正向相互作用,从而促进了凋落物的分解和再循环,增加自身α多样性[53].
NMDS分析结果表明,不同的植物叶片附着细菌群落之间相似度较低(图 4),ANOSIM分析结果(表 2)也显示它们之间均存在显著差异. 外界环境和植物本身都对附着细菌群落造成一定的影响,水体中的化学物质,包括氮磷营养盐、溶解性有机物等,包括植物叶片分泌或渗出的物质,比如糖类、有机酸、酚类等[54],它们作为细菌生长的营养物质或者干扰物质,都会对细菌群落结构造成影响[55]. 例如,随着植物物种多样性升高,体系NH3-N水平下降会导致叶片的可溶性糖含量增加和可溶性氨基酸含量减少[56],从而潜在地影响叶表面的生化特性. 关于植物种类对附着细菌群落结构影响的研究发现:宿主专一性对附着细菌群落有重要影响,不同种类的植物叶片由于其独特的化学组成、叶片渗出物、物理结构等因素,具有差异显著的附着细菌群落[10, 57]. 本研究结果显示,微齿眼子菜叶片附着细菌群落的β多样性显著更高. 这可能是两方面因素导致的,一方面是因为微齿眼子菜叶片结构较复杂,表面可能存在更多的微环境异质性[58]. 另一方面可能是由于微齿眼子菜的生长代谢更易受到环境因子的影响,进而影响附着细菌[59]. 微齿眼子菜本身对环境条件的变化比苦草、黑藻等沉水植物更加敏感[60],环境的变化使其叶表有更强的异质性,其叶表附着细菌群落会有更高的β多样性. 此外,水体环境因子如氨氮、硝氮对附着细菌群落结构的影响都非常大(图 5,6),氮素对沉水植物叶片生物膜细菌群落具有重要影响[61]. 水体氨氮、硝态氮等不同形态氮素浓度的变化可能会影响沉水植物叶片分泌物的组成及含量,从而影响附着细菌的群落结构[62];同时它们与附着细菌生物膜中反硝化细菌群落的变化显著相关[63]. 不同沉水植物叶片附着细菌群落差异的具体机制仍然需要更深入的研究探讨.
在门分类水平上,不同植物的叶片附着细菌丰度不同,苦草叶片中拟杆菌门、β-变形菌纲和γ-变形菌纲较多,微齿眼子菜和黑藻以α-变形菌纲和拟杆菌门居多,穗状狐尾藻中α-变形菌纲、β-变形菌纲、拟杆菌门所占比例较高(图 3). 几种沉水植物的叶片形态差异极大,苦草叶片为线型;微齿眼子菜、黑藻和穗状狐尾藻叶片为披针形,边缘呈浅波状,不同的叶形为细菌提供了不同的附着基质[64]. 不同的植物有不同种类的内生细菌[45],在生长过程中植物不同的内生细菌可能会通过组织缝隙,表面裂口等物理通道或随植物的代谢产物一并迁移到植物叶表,最终各自形成独特的细菌群落结构[9]. 此外,叶片附着细菌的生长繁殖可能与周围水体环境密切相关,营养盐负荷、特殊的生境条件等都会影响叶片附着细菌的种类和丰度[64].
3.3 不同植物叶片附着细菌群落网络的差异生态网络可以代表生态系统中的各种生物相互作用,例如捕食、竞争和共生[65]. 物种(节点)通过相互作用成对连接[53],网络拓扑属性为理解细菌群落内成员的潜在相互作用或生态位分化提供了线索[66]. 本研究构建了不同植物叶片附着细菌的物种共现网络预测这些群落中物种之间的生态相互作用. 注意到高植物物种多样性体系中所有植物的网络有最高的平均度,表明形成网络的类群之间有积极的联系;正相关关系接近负相关关系的2倍(表 3),可能意味着各种植物叶片的附着细菌的成员在附生群落中具有更强烈的积极相互作用[67]. 同时,研究发现穗状狐尾藻叶片附着细菌网络中负相关关系比例较高(49.5 %),这表明物种之间的竞争较强,网络在干扰下更加稳定[68]. 苦草叶片附着细菌网络有最高的模块性(表 3),显示苦草叶片中附着细菌网络更复杂[69]. 在一项群落演替研究中证实,捕食者和猎物之间的相互作用强度及其复杂性有助于食物网的稳定性[65, 70]. 随着网络中更高程度和更积极的联系,叶片附着细菌的成员可能在附生群落中具有更强烈的相互促进作用. 附生细菌含有许多参与植物叶片生物膜形成的基因,这需要不同种类细菌的合作[70],并且可能在附生网络中形成较高比例的正相关关系. 特别是黑藻和苦草叶片附生细菌网络中存在更多的正相关关系,这意味着它们的叶片附着细菌之间有更强共生关系,可能对不同的环境有更强的生态适应能力[71].
网络分析还可以揭示关键物种的信息,一般来说如模块核心和连接点等高连接度的节点由于对细菌群落结构的重要影响而被视为关键物种[29],因为它们在维持细菌群落组成和功能的稳定性方面发挥着重要作用[72]. 纤维素是植物细胞壁的重要组成部分,植物叶片的组成和分泌物中富含多糖和纤维素,变形菌、拟杆菌在多糖或木质纤维素降解中发挥重要作用[69],这可以解释各植物叶片附着细菌网络关键物种都出现了变形菌门和拟杆菌门. 植物叶片生物膜在构建过程中和一些附着细菌有着密不可分的关系,黑藻叶片附着细菌网络关键物种中OTU160584和OTU101083属于嗜甲基菌(g_Methylophilus),穗状狐尾藻和微齿眼子菜叶片附着细菌网络的关键物种中OTU85983和OTU131840属于红杆菌(f_Rhodobacter),它们都在叶片生物膜的形成中发挥关键作用[71, 73]. 这些OTU可能为其它细菌在构建叶片生物膜时提供碳养分,从而影响整个附生细菌群落在植物叶片上的构建[70]. 苦草和黑藻附着细菌网络的关键物种中OTU185、OTU115144和OTU109577属于黄杆菌属(g_Flavobacterium),这一快生型细菌被称为营养盐(磷酸盐)增溶剂,而磷在DNA复制和转录中起重要作用,在限磷条件下,快速生长的细菌尤其受到影响[74]. 在植物分解阶段磷含量的快速损失为黄杆菌与其它细菌之间的优先合作提供了机会,促进参与分解的细菌群落之间的合作[52]. 可以推测,这些细菌可能在植物叶片生物膜的构建、糖类的降解等过程中发挥着至关重要的作用.
4 结论1) 浮游细菌和叶片附着细菌的群落多样性和结构具有显著差异. 与浮游细菌群落相比,叶片附着细菌群落表现出较高的α多样性(OTU丰富度和系统发育多样性). 植物可通过分泌物或光合产物提供养分来支持更高多样性的细菌群落,同时为细菌提供更多的生存空间.
2) 与单一植物物种体系相比,高植物物种多样性体系中植物叶片附着细菌群落α多样性较高. 更多种类的沉水植物能提供更多种类的营养物质,从而可支持更多的细菌物种存活.
3) 沉水植物叶片附着细菌群落优势菌群属于变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes). 不同植物叶片附着细菌群落结构存在差异,宿主选择效应是塑造沉水植物叶片附着细菌群落的重要因子. 植物也通过影响水体中的氮、磷营养盐等浓度,间接影响叶片附着细菌群落的多样性和结构.
4) 高植物物种多样性体系中苦草叶片附着细菌网络最复杂,穗状狐尾藻叶片附着细菌网络中物种之间竞争较强,网络更加稳定. 嗜甲基菌、红杆菌、黄杆菌是物种共现网络的关键物种,可能在植物叶片生物膜的构建、糖类的降解等过程中发挥关键作用.
| [1] |
Liu Y, Fu WL, Cao Y et al. Study on the functional traits of submerged macrophytes. Plant Science Journal, 2017, 35(3): 444-451. [刘洋, 付文龙, 操瑜等. 沉水植物功能性状研究的思考. 植物科学学报, 2017, 35(3): 444-451. DOI:10.11913/PSJ.2095-0837.2017.30444] |
| [2] |
Zhang SH, Zhou TT, Liu YS. Characteristics of microbial communities in biofilm of four aquatic macrophytes in Xinkai River. Water Resources Protection, 2020, 36(3): 83-88, 104. [张松贺, 周甜甜, 刘远思. 新开河4种水生植物表面附着微生物群落特征. 水资源保护, 2020, 36(3): 83-88, 104.] |
| [3] |
Lv XY, Zhang SH, Liu KH et al. Advances in ecological function and interaction mechanism of aquatic macrophyte-biofilm system. Water Resources Protection, 2015, 31(2): 20-25. [吕小央, 张松贺, 刘凯辉等. 水生植物-生物膜体系的生态功能与互作机制研究进展. 水资源保护, 2015, 31(2): 20-25.] |
| [4] |
Gordon-Bradley N, Lymperopoulou DS, Williams HN. Differences in bacterial community structure on Hydrilla verticillata and Vallisneria americana in a freshwater spring. Microbes and Environments, 2014, 29(1): 67-73. DOI:10.1264/jsme2.me13064 |
| [5] |
Ma J, Fan Z, Wang GX et al. Diversity and community structure of epiphytic bacteria on different submerged macrophytes in Lake Hongze. J Lake Sci, 2016, 28(4): 852-858. [马杰, 范婤, 王国祥等. 洪泽湖3种沉水植物附着细菌群落结构及多样性. 湖泊科学, 2016, 28(4): 852-858. DOI:10.18307/2016.0419] |
| [6] |
Zhao DY, Liu P, Fang C et al. Submerged macrophytes modify bacterial community composition in sediments in a large, shallow, freshwater lake. Canadian Journal of Microbiology, 2013, 59(4): 237-244. DOI:10.1139/cjm-2012-0554 |
| [7] |
He D, Ren LJ, Xing P et al. Research progress of diversity and community structure of epiphytic bacteria on submerged macrophytes. Chinese Bulletin of Life Sciences, 2014, 26(2): 161-168. [何聃, 任丽娟, 邢鹏等. 沉水植物附着细菌群落结构及其多样性研究进展. 生命科学, 2014, 26(2): 161-168. DOI:10.13376/j.cbls/2014025] |
| [8] |
Wu QL, Xing P, Li HB et al. Impacts of regime shift between phytoplankton and macrophyte on the microbial community structure and its carbon cycling in lakes. Microbiology China, 2013, 40(1): 87-97. [吴庆龙, 邢鹏, 李化炳等. 草藻型稳态转换对湖泊微生物结构及其碳循环功能的影响. 微生物学通报, 2013, 40(1): 87-97. DOI:10.13344/j.microbiol.china.2013.01.003] |
| [9] |
Bellés-Garulera J, Vila M, Borrull E et al. Variability of planktonic and epiphytic vibrios in a coastal environment affected by Ostreopsis blooms. Scientia Marina, 2016, 80(S1): 97-106. DOI:10.3989/scimar.04405.01a |
| [10] |
He D, Ren LJ, Wu QL. Epiphytic bacterial communities on two common submerged macrophytes in Taihu Lake: Diversity and host-specificity. Chinese Journal of Oceanology and Limnology, 2012, 30(2): 237-247. DOI:10.1007/s00343-012-1084-0 |
| [11] |
Zeng J, Bian YQ, Xing P et al. Macrophyte species drive the variation of bacterioplankton community composition in a shallow freshwater lake. Applied and Environmental Microbiology, 2012, 78(1): 177-184. DOI:10.1128/AEM.05117-11 |
| [12] |
Yang F, Yin J, Zhang YM et al. Research on characteristics of epiphytic organism of two submerged macrophytes and its response to water depth. Acta Scientiae Circumstantiae, 2018, 38(6): 2427-2434. [杨飞, 尹杰, 张毅敏等. 两种沉水植物附着生物种群特征对水深的响应研究. 环境科学学报, 2018, 38(6): 2427-2434. DOI:10.13671/j.hjkxxb.2017.0501] |
| [13] |
He D, Ren LJ, Wu QL. Contrasting diversity of epibiotic bacteria and surrounding bacterioplankton of a common submerged macrophyte, Potamogeton crispus, in freshwater lakes. FEMS Microbiology Ecology, 2014, 90(3): 551-562. DOI:10.1111/1574-6941.12414 |
| [14] |
Casartelli MR, Ferragut C. Influence of seasonality and rooted aquatic macrophyte on periphytic algal community on artificial substratum in a shallow tropical reservoir. International Review of Hydrobiology, 2015, 100(5/6): 158-168. DOI:10.1002/iroh.201401773 |
| [15] |
Liu KH, Zhang SH, Lv XY et al. The characteristics of epiphytic microbes of three submerged macrophytes in Lake Hua-Shen. J Lake Sci, 2015, 27(1): 103-112. [刘凯辉, 张松贺, 吕小央等. 南京花神湖3种沉水植物表面附着微生物群落特征. 湖泊科学, 2015, 27(1): 103-112. DOI:10.18307/2015.0113] |
| [16] |
Cai XL, Gao G, Tang XM et al. The response of epiphytic microbes to habitat and growth status of Potamogeton malaianus Miq. in Lake Taihu. Journal of Basic Microbiology, 2013, 53(10): 828-837. DOI:10.1002/jobm.201200220 |
| [17] |
Zhao DY, Cao XY, Zeng J et al. Effects of macrobenthic bioturbation on the abundance and community composition of ammonia-oxidizing prokaryotes under different temperatures. Journal of Freshwater Ecology, 2017, 32(1): 405-414. DOI:10.1080/02705060.2017.1320307 |
| [18] |
Xu HM, Zhao DY, Zeng J et al. Distinct successional patterns and processes of free-living and particle-attached bacterial communities throughout a phytoplankton bloom. Freshwater Biology, 2020, 65(8): 1363-1375. DOI:10.1111/fwb.13505 |
| [19] |
He RJ, Zeng J, Zhao DY et al. Contrasting patterns in diversity and community assembly of Phragmites australis root-associated bacterial communities from different seasons. Applied and Environmental Microbiology, 2020, 86(14): e00379-e00320. DOI:10.1128/AEM.00379-20 |
| [20] |
Zhao DY, Xu HM, Zeng J et al. Community composition and assembly processes of the free-living and particle-attached bacteria in Taihu Lake. FEMS Microbiology Ecology, 2017, 93(6): fix062. DOI:10.1093/femsec/fix062 |
| [21] |
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [22] |
Edgar RC, Haas BJ, Clemente JC et al. UCHIME improves sensitivity and speed of chimera detection. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [23] |
Schloss PD. The effects of alignment quality, distance calculation method, sequence filtering, and region on the analysis of 16S rRNA gene-based studies. PLoS Computational Biology, 2010, 6(7): e1000844. DOI:10.1371/journal.pcbi.1000844 |
| [24] |
Wang Q, Garrity GM, Tiedje JM et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [25] |
Bokulich NA, Subramanian S, Faith JJ et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing. Nature Methods: Techniques for Life Scientists and Chemists, 2013, 10(1). DOI:10.1038/nmeth.2276 |
| [26] |
Caporaso JG, Kuczynski J, Stombaugh J et al. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods: Techniques for Life Scientists and Chemists, 2010, 7(5). DOI:10.1038/nmeth.f.303 |
| [27] |
Liu L, Chen H, Liu M et al. Response of the eukaryotic plankton community to the cyanobacterial biomass cycle over 6 years in two subtropical reservoirs. The ISME Journal, 2019, 13(9): 2196-2208. DOI:10.1038/s41396-019-0417-9 |
| [28] |
Liu LM, Wang SS, Ji JN et al. Characteristics of microbial eukaryotic community recovery in eutrophic water by using ecological floating beds. Science of the Total Environment, 2020, 711: 134551. DOI:10.1016/j.scitotenv.2019.134551 |
| [29] |
Jiao CC, Zhao DY, Zeng J et al. Disentangling the seasonal co-occurrence patterns and ecological stochasticity of planktonic and benthic bacterial communities within multiple lakes. Science of the Total Environment, 2020, 740: 140010. DOI:10.1016/j.scitotenv.2020.140010 |
| [30] |
Guimerà R, Nunes Amaral LA. Functional cartography of complex metabolic networks. Nature, 2005, 433(7028). |
| [31] |
Zhang B, Ning DL, Yang YF et al. Biodegradability of wastewater determines microbial assembly mechanisms in full-scale wastewater treatment plants. Water Research, 2020, 169: 115276. DOI:10.1016/j.watres.2019.115276 |
| [32] |
Csárdi G, Nepusz T. The igraph software package for complex network research. Journal of Systems Science & Complexity, 2006, 1695: 1-9. |
| [33] |
Zhao DY, Shen F, Zeng J et al. Network analysis reveals seasonal variation of co-occurrence correlations between Cyanobacteria and other bacterioplankton. Science of the Total Environment, 2016, 573: 817-825. DOI:10.1016/j.scitotenv.2016.08.150 |
| [34] |
Kozich JJ, Westcott SL, Baxter NT et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied and Environmental Microbiology, 2013, 79(17): 5112-5120. DOI:10.1128/AEM.01043-13 |
| [35] |
Caporaso JG, Kuczynski J, Stombaugh J et al. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [36] |
Dixon P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science, 2003, 14(6): 927-930. DOI:10.1111/j.1654-1103.2003.tb02228.x |
| [37] |
Zhang WZ, Wang L, Chen L et al. Proliferation of filamentous green algae along with submerged macrophytes planting, and the role of microbe. Ecological Engineering, 2019, 139: 105570. DOI:10.1016/j.ecoleng.2019.07.040 |
| [38] |
Cadotte MW, Cavender-Bares J, Tilman D et al. Using phylogenetic, functional and trait diversity to understand patterns of plant community productivity. PLoS One, 2009, 4(5): e5695. DOI:10.1371/journal.pone.0005695 |
| [39] |
Beattie GA, Lindow SE. The secret life of foliar bacterial pathogens on leaves. Annual Review of Phytopathology, 1995, 33: 145-172. DOI:10.1146/annurev.py.33.090195.001045 |
| [40] |
Wright DH. Species-energy theory: An extension of species-area theory. Oikos, 1983, 41(3): 496. DOI:10.2307/3544109 |
| [41] |
Liu HW, Brettell LE, Singh B. Linking the phyllosphere microbiome to plant health. Trends in Plant Science, 2020, 25(9): 841-844. DOI:10.1016/j.tplants.2020.06.003 |
| [42] |
Gong TY, Xin XF. Phyllosphere microbiota: Community dynamics and its interaction with plant hosts. Journal of Integrative Plant Biology, 2021, 63(2): 297-304. DOI:10.1111/jipb.13060 |
| [43] |
Hempel M, Blume M, Blindow I et al. Epiphytic bacterial community composition on two common submerged macrophytes in brackish water and freshwater. BMC Microbiology, 2008, 8: 58. DOI:10.1186/1471-2180-8-58 |
| [44] |
Zhao BY, Xing P, Wu QL. Microbes participated in macrophyte leaf litters decomposition in freshwater habitat. FEMS Microbiology Ecology, 2017, 93(10): fix108. DOI:10.1093/femsec/fix108 |
| [45] |
Xiong C, Zhu YG, Wang JT et al. Host selection shapes crop microbiome assembly and network complexity. New Phytologist, 2021, 229(2): 1091-1104. DOI:10.1111/nph.16890 |
| [46] |
Lu SJ, He RJ, Zhao DY et al. Effects of shading levels on the composition and co-occurrence patterns of bacterioplankton and epibiotic bacterial communities of Cabomba caroliniana. Science of the Total Environment, 2021, 785: 147286. DOI:10.1016/j.scitotenv.2021.147286 |
| [47] |
van der Gucht K, Vandekerckhove T, Vloemans N et al. Characterization of bacterial communities in four freshwater lakes differing in nutrient load and food web structure. FEMS Microbiology Ecology, 2005, 53(2): 205-220. DOI:10.1016/j.femsec.2004.12.006 |
| [48] |
Li LL, Tang XM, Gao G et al. Influence of submerged vegetation restoration on bacterial diversity and community composition in West Lake. J Lake Sci, 2013, 25(2): 188-198. [李琳琳, 汤祥明, 高光等. 沉水植物生态修复对西湖细菌多样性及群落结构的影响. 湖泊科学, 2013, 25(2): 188-198. DOI:10.18307/2013.0203] |
| [49] |
Hahn MW, Kasalický V, Jezbera J et al. Limnohabitans curvus gen. nov., sp. nov., a planktonic bacterium isolated from a freshwater lake. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(Pt 6): 1358-1365. DOI:10.1099/ijs.0.013292-0 |
| [50] |
Bian GG, Zhao WD, Da L. Thea research and applicacian of the allelopathic effects of submerged plants inhibit the growth of algae. Northern Environment, 2012, 24(1): 59-64. [边归国, 赵卫东, 达来. 沉水植物化感作用抑制藻类生长的研究与应用. 北方环境, 2012, 24(1): 59-64.] |
| [51] |
Lindow SE, Brandl MT. Microbiology of the phyllosphere. Applied and Environmental Microbiology, 2003, 69(4): 1875-1883. DOI:10.1128/aem.69.4.1875-1883.2003 |
| [52] |
Xiao X, Lou LP, Li H et al. Algal control ability of allelopathically active submerged macrophytes: A review. Chinese Journal of Applied Ecology, 2009, 20(3): 705-712. [肖溪, 楼莉萍, 李华等. 沉水植物化感作用控藻能力评述. 应用生态学报, 2009, 20(3): 705-712.] |
| [53] |
Zhao BY, Xing P, Wu QL. Interactions between bacteria and fungi in macrophyte leaf litter decomposition. Environmental Microbiology, 2021, 23(2): 1130-1144. DOI:10.1111/1462-2920.15261 |
| [54] |
Fiehn C. Methotrexate transport mechanisms: the basis for targeted drug delivery and ß-folate-receptor-specific treatment. Clinical and Experimental Rheumatology, 2010, 28(5 Suppl61). |
| [55] |
Tuomisto H, Ruokolainen K. Analyzing or explaining beta diversity? Understanding the targets of different methods of analysis. Ecology, 2006, 87(11): 2697-2708. DOI:10.1890/0012-9658(2006)87[2697:aoebdu]2.0.co;2 |
| [56] |
Cao T, Ni LY, Xie P. Acute biochemical responses of a submersed macrophyte, Potamogeton crispus L., to high ammonium in an aquarium experiment. Journal of Freshwater Ecology, 2004, 19(2): 279-284. DOI:10.1080/02705060.2004.9664542 |
| [57] |
Lachnit T, Meske D, Wahl M et al. Epibacterial community patterns on marine macroalgae are host-specific but temporally variable. Environmental Microbiology, 2011, 13(3): 655-665. DOI:10.1111/j.1462-2920.2010.02371.x |
| [58] |
Li CL, Chang MY, Dong J et al. Responses of eutrophic ponds to Ceratophyllum demersum and Potamogeton pectinatus. Progress in Fishery Sciences, 2020, 41(4): 125-133. [李晨露, 常孟阳, 董静等. 富藻养殖水体中金鱼藻和篦齿眼子菜的抑藻效果研究. 渔业科学进展, 2020, 41(4): 125-133.] |
| [59] |
Jeppesen E, Jensen JP, Søndergaard M et al. Top-down control in freshwater lakes: The role of nutrient state, submerged macrophytes and water depth. Hydrobiologia, 1997, 342/343: 151-164. DOI:10.1023/A:1017046130329 |
| [60] |
Zhu DT. Effects of light, temperature and nitrogen on the growth of three kinds of aquatic plants[Dissertation]. Jinhua: Zhejiang Normal University, 2011. [朱丹婷. 光照强度、温度和总氮浓度对三种沉水植物生长的影响[学位论文]. 金华: 浙江师范大学, 2011. ]
|
| [61] |
Yan LY, Zhang SH, Lin D et al. Nitrogen loading affects microbes, nitrifiers and denitrifiers attached to submerged macrophyte in constructed wetlands. Science of the Total Environment, 2018, 622/623: 121-126. DOI:10.1016/j.scitotenv.2017.11.234 |
| [62] |
Yan LL, Xiao YM, Bing H et al. Ammonium loading disturbed the microbial food webs in biofilms attached to submersed macrophyte Vallisneria natans. Science of the Total Environment, 2019, 659: 691-698. DOI:10.1016/j.scitotenv.2018.12.423 |
| [63] |
Yan DB, Xia PH, Song X et al. Community structure and functional diversity of epiphytic bacteria and planktonic bacteria on submerged macrophytes in Caohai Lake, southwest of China. Annals of Microbiology, 2019, 69(9): 933-944. DOI:10.1007/s13213-019-01485-4 |
| [64] |
Ma J. Characteristics of bacterial community structure attached to typical submerged plants in shallow lakes[Dissertation]. Nanjing: Nanjing Normal University, 2016. [马杰. 浅水湖泊典型沉植物附着细菌群落结构特征[学位论文]. 南京: 南京师范大学, 2016. ]
|
| [65] |
Neutel AM, Heesterbeek JAP, van de Koppel J et al. Reconciling complexity with stability in naturally assembling food webs. Nature, 2009, 458(7234): 110. DOI:10.1038/nature06154 |
| [66] |
Cumming GS, Bodin Ö, Ernstson H et al. Network analysis in conservation biogeography: Challenges and opportunities. Diversity and Distributions, 2010, 16(3): 414-425. DOI:10.1111/j.1472-4642.2010.00651.x |
| [67] |
He D, Zheng JW, Ren LJ et al. Substrate type and plant phenolics influence epiphytic bacterial assembly during short-term succession. Science of the Total Environment, 2021, 792: 148410. DOI:10.1016/j.scitotenv.2021.148410 |
| [68] |
Hu SW, He RJ, Wang WJ et al. Composition and co-occurrence patterns of Phragmites australis rhizosphere bacterial community. Aquatic Ecology, 2021, 55(2): 695-710. DOI:10.1007/s10452-021-09855-4 |
| [69] |
Si TT, Chen HZ, Qiu Z et al. Bacterial succession in epiphytic biofilms and deciduous layer sediments during Hydrilla verticillata decay: A field investigation. Journal of Environmental Sciences, 2020, 93: 193-201. DOI:10.1016/j.jes.2020.03.008 |
| [70] |
Kampf J, Gerwig J, Kruse K et al. Selective pressure for biofilm formation in Bacillus subtilis: Differential effect of mutations in the master regulator SinR on bistability. mBio, 2018, 9(5): e01464-e01418. DOI:10.1128/mBio.01464-18 |
| [71] |
He D, Ren LJ, Wu QL. Growing season drives the compositional changes and assembly processes of epiphytic bacterial communitiesof two submerged macrophytes in Taihu Lake. FEMS Microbiology Ecology, 2020, 96(4): fiaa025. DOI:10.1093/femsec/fiaa025 |
| [72] |
Sun XJ, Cao XY, Zhao DY et al. The pattern of sedimentary bacterial communities varies with latitude within a large eutrophic lake. Limnologica, 2021, 87: 125860. DOI:10.1016/j.limno.2021.125860 |
| [73] |
Elifantz H, Horn G, Ayon M et al. Rhodobacteraceae are the key members of the microbial community of the initial biofilm formed in Eastern Mediterranean coastal seawater. FEMS Microbiology Ecology, 2013, 85(2): 348-357. DOI:10.1111/1574-6941.12122 |
| [74] |
Wu L, Qin HY, Zhu MY et al. Distribution characteristics and controlling factors of nitrifying microorganisms in freshwater and sediment of eutrophic zones in Lake Taihu in autumn. J Lake Sci, 2017, 29(6): 1312-1323. [吴玲, 秦红益, 朱梦圆等. 太湖富营养化湖区秋季水体和沉积物中硝化微生物分布特征及控制因素. 湖泊科学, 2017, 29(6): 1312-1323. DOI:10.18307/2017.0603] |
 2022, Vol. 34
2022, Vol. 34 

