(2: 南昌大学鄱阳湖环境与资源利用教育部重点实验室, 南昌 330031)
(3: 江西省流域生态演变与生物多样性重点实验室, 南昌 330031)
(4: 江西省水生生物保护救助中心, 南昌 330096)
(2: Ministry of Education, Key Laboratory of Environment and Resource Utilization of Poyang Lake, Nanchang University, Nanchang 330031, P. R. China)
(3: Jiangxi Province Key Laboratory of Watershed Ecosystem Change and Biodiversity, Nanchang 330031, P. R. China)
(4: Jiangxi Aquatic Animal Protection and Rescue Center, Nanchang 330096, P. R. China)
淡水生态系统是人类社会和经济发展的基础,人类活动的直接影响导致淡水生物多样性的下降速度超过陆地和海洋生态系统[1]。20世纪淡水鱼的灭绝速度超过了所有其他脊椎动物群体[2]。鱼类多样性是水生生物多样性的重要组成部分,鱼类多样性的保护对水生生态系统的健康和稳定具有重要意义[3]。鱼类物种多样性是目前监测水生生态系统健康状况的关键指标,具有重要的生态价值[4]。为了调查特定区域鱼类的物种数量和物种组成,通常需要进行实地调查,利用直接捕获的抽样方法或水下目视普查[5]。然而,这种野外调查是劳动密集型的、耗时的、昂贵的,并且需要高度专业的物种鉴定分类知识[6]。
在过去的十年中,环境DNA宏条形码作为生物多样性监测中推断物种存在的间接遗传标记受到越来越多的关注[7-8]。环境DNA宏条形码技术通过直接提取环境样品中的DNA,运用PCR扩增和二代测序技术对目标物种进行精准识别,其操作过程不需收集目标生物个体, 具有非破坏性、简易和灵敏等特点,弥补了传统形态学监测的不足[6]。环境DNA宏条形码技术已被证明是一种灵敏且经济有效的淡水群落调查方法[9-11]。与其他调查方法的比较表明,环境DNA宏条形码数据通常会增加物种的检出和提高物种丰富度估计[8]。在我国全面实施长江“10年禁渔”期间,许多传统水生生物监测方法难以适应新的监测要求[12]。相比传统鱼类监测方法,环境DNA宏条形码在鱼类多样性评估及生态保护方面具有明显的优势[13]。
赣江位于长江中下游南岸,是鄱阳湖最大的入湖河流,是江西省最大的河流,也是江西水运的重要承担者[14-15]。赣江是鄱阳湖最大的入湖河流,鱼类资源丰富,是优质的产卵繁殖、洄游区域[16]。但是由于人类活动的干扰,赣江鱼类的生存受到严重威胁[17]。此外,赣江下游航运发达,为传统方法的采样也带来了一定的难度,因此,迫切需要对环境无破坏、对鱼类生存无威胁、取样方便省力的方法进行物种资源的评估。
在利用环境DNA宏条形码技术进行鱼类资源的监测时,针对采集样品的位置存在一些疑虑:根据鱼类栖息水层的不同,是否需要分层取样?由于环境DNA在水中随水流动聚集,是否需要近岸和离岸分别取样?许多研究者已针对这些疑虑进行了探索。在加拿大的一个实验湖泊中,作者在每个样点取6个不同深度的水样,得出每个样点物种的相对序列比例由于深度的不同而不同,在湖水存在热分层现象时,鱼类的群落组成也不同等结论[18]。也有研究者在冬季的近岸区域检测到了湖体中心较深处的物种环境DNA[19]。但是由于鱼类对环境的适应,在不同的水域调查可能会出现不同的结果。
本研究旨在利用环境DNA宏条形码技术对赣江下游南昌段的鱼类多样性进行评估,探索不同季节、不同水层以及近岸和离岸的鱼类组成及群落结构。以期为后续赣江鱼类资源环境DNA宏条形码监测实施不同目的的采样策略提供依据,为使用环境DNA宏条形码技术研究流水系统鱼类多样性提供技术参考。
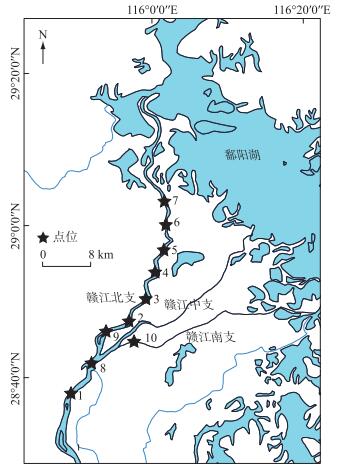
1 材料与方法 1.1 环境DNA样品的采集及处理于夏季(2020年7月)、秋季(2020年10月)、冬季(2021年1月)和春季(2021年5月)在赣江下游南昌段进行环境DNA样品的采集,包括近岸7个样点和离岸3个样点(样点8、9和10)(图 1)。近岸7个样点进行了4个季节的采集,共28个样本,离岸3个样点只采集了秋季和春季(因离岸样点的采集需要船只,而禁渔执行期内难以获得渔政执法部门的批准,所以仅采了2个季节),且离岸3个样点在秋季分上、中、下3个水层进行采集,共9个样本,而春季未进行分层采样(因为在赣江秋季的分层样本中和鄱阳湖的分层样本中均显示鱼类群落结构不具分层差异),因而共采集了40个样本。在近岸处(无需乘船)使用自制的长手柄采水器采集1 L水于已灭菌的广口瓶中,立即置于冰桶中保存或运输。在离岸处乘坐快艇使用1 L采水器分别采集上层水样(河水表面以下0.5~1 m处)、中层水样(水深的一半)、下层水样(河底部以上0.5~1 m处)于无菌广口瓶中。离岸处3个样点的水深情况为:秋季样点8、9、10的水深分别为7.2、7.0、7.5 m;春季样点8、9、10的水深分别为13.5、12.2、14.0 m。所有水样冰上保存,且均在4 h内使用无菌的0.45 μm混合纤维素滤膜进行真空电动泵抽滤。为了防止在抽滤过程中的DNA污染,每次抽滤均设置1个空白对照。为了减少误差,整个水样采集、过滤过程均由同一个受过专业培训的研究生完成,且全程戴无菌手套。抽滤完成之后,滤膜保存于-20℃。使用多参数水质仪测量水深、水温(Tem)、酸碱度(pH)、化学需氧量(COD)、叶绿素a(Chl.a)和溶解氧(DO)。
|
图 1 赣江采样点位图 Fig.1 Distribution of water sampling locations in Ganjiang River |
为确保滤膜DNA的提取过程无污染,在每批提取时使用1张未使用过的滤膜作为DNA提取空白对照。采用DNeasyⓇBlood&Tissue Kit试剂盒(QIAGEN,德国)进行滤膜DNA的提取(包括抽滤时的空白对照和提取DNA时的空白对照),最后用60 μL Elution Buffer洗脱DNA,保存于-20℃冰箱。使用Qubit(Thermo Fisher Scientific,中国)测量DNA浓度。利用通用引物Teleo02:AAACTCGTGCCAGCCAC,GGGTATCTAATCCCAGTTTG[20]针对脊椎动物12S区域(扩增长度约167 bp)进行PCR扩增,并设置阴性对照,确保过程中未受到污染。引物前添加了样本特异性的Barcode序列。每份样品重复扩增3份,并设置阴性对照。反应总体积20 μL,由Q5 High-Fidelity DNA Polymerase(2000 U/mL)(New England Biolabs,USA)0.2 μL,5×reaction buffer和5×GC buffer各4 μL,dNTP mixture(2.5 mmol/L)1.6 μL,10 μmol/L的正反引物各0.8 μL,DNA模板3 μL及灭菌双蒸水组成。使用2 % 的凝胶电泳对PCR产物进行检测,PCR阴性对照和空白对照的样本均未出现扩增条带,表明在采样及实验操作过程中均未产生污染。PCR程序如下:95℃预变性1 min之后进入35个循环(95℃变性30 s,55℃退火30 s,72℃延伸45 s),72℃再延伸10 min。
1.3 高通量测序将上述DNA样本送至上海凌恩生物科技有限公司进行高通量测序,样本DNA片段在Illumina HiSeq 2500平台进行测序。采用denovo和reference结合的方式去除嵌合体。按照97 % 相似性进行OTU聚类,聚类过程中去除嵌合体之后即可获得OTU的代表序列(使用降噪算法生成代表序列,如dada2的ASV或usearch的zOTU在物种注释时会更准确。ASV聚类是代表未来的分析方法标准,它优于OTU聚类结果,比OTU聚类分析结果更准确)。物种比对注释用的是基于nt筛选的鱼类参考序列数据库。
1.4 数据分析将上述测序后生成的OTU分类注释表进行以下处理以确定每个OTU对应的物种:首先,去除表中的非鱼序列;其次,删除相似度小于98 %、序列覆盖度小于100 % 的序列;接着,根据赣江历史记录鱼类的参考数据库与公司结果核对剩余的序列信息,在赣江历史记录鱼类数据库中Blast之后,出现同一OTU对应多种鱼的OTU的情况,如果公司的结果相似度大于98 %,序列覆盖度为100 %,且仅对应一种鱼,则取该结果。本研究的数据未出现反之的情况。最后,保证确定的物种Max Score排列在第一位。
根据《中国动物志-硬骨鱼纲-鲤形目》中卷[21]、《中国动物志-硬骨鱼纲-鲤形目》下卷[22]、《中国淡水鱼类检索》[23]以Fish of the Word,Fifth Edition和Fishbase(www.fishbase.se)[17]校对鱼类物种的名称和分类系统。根据生活习性将鱼类划分为5种类型:河海洄游型(RM)、江(河)湖洄游型(MRL)、河流(山溪)型(RT)、湖泊定居型(LT)[24-25]。刀鲚定为江海洄游型进行数据分析[26]。
对不同季节、不同水层、不同位置的物种数量进行正态检验,使用配对样本Wilcoxon符号秩检验对不同季节、不同水层、不同位置的物种数量进行差异性检验。
利用Chao 1(Chao1 index)指数来反映群落的丰富度,香农指数(Shannon index,H′)来反映群落的多样性程度,用Pielou均匀度指数来评估群落内各物种分布的均匀程度。使用非度量多维尺度分析(non-metric multidimensional scaling analysis;NMDS)来比较样本之间的差异性或相似性。并进一步用相似性分析(analysis of similarities;ANOSIM)来检验组间差异是否显著大于组内差异。使用冗余分析(redundancy analysis;RDA)进行环境因子关联分析。利用相关性Heatmap图呈现群落与环境因子之间相关性系数。
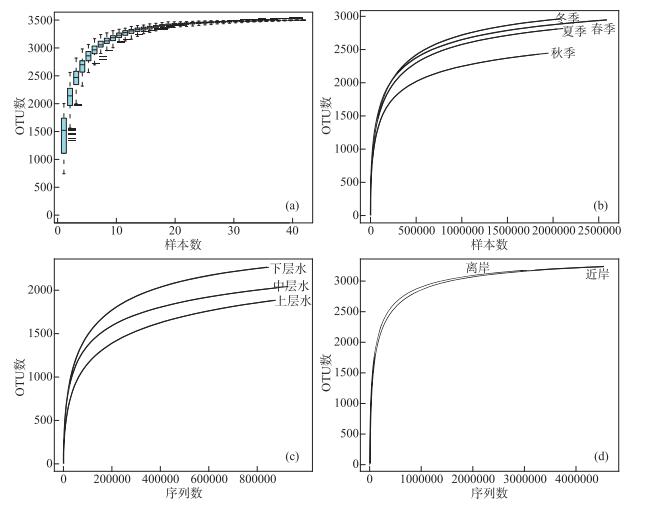
2 结果 2.1 基于环境DNA宏条形码技术的测序结果本研究基于环境DNA宏条形码技术监测共得到有效拼接序列11931367条,平均序列长度171 bp。将有效序列数按照≥97 % 的序列相似度进行聚类,共得到3498个鱼类OTUs。赣江鱼类调查样本的累积曲线和稀释曲线如图 2。图 2a中,当样本数约为20时,曲线趋于平缓势,随着样本数的增加,观察到的OTU数目不再明显增多,表明样本数足够。在季节(图 2b)、水层(图 2c)、近岸离岸(图 2d)分组时的稀释曲线表明测序深度满足要求。
|
图 2 赣江鱼类样本累积曲线和稀释曲线 Fig.2 Accumulation curve and dilution curve of Ganjiang River fish |
本次调查共检测到鱼类114种(附表Ⅰ),包括赣江历史记录种83种,赣江新记录种31种[16, 27-29]。在种类上,鲤形目最多,有67种,其次是鲇形目15种,鲈形目15种。在时间上,春季鉴定到103种,夏季100种,秋季69种,冬季鉴定到90种。仅在春季被检测到鱼类有铜鱼、贝氏䱗、纵带鮠、长吻鮠、瓦氏黄颡鱼、Spinibarbus sp.。短臀白鱼和黄鳝仅在夏季被检测到。莫桑比克罗非鱼、小头高原鱼和大盖巨脂鲤仅在秋季被检测到。春季、夏季和冬季的物种数极显著多于秋季(P < 0.001),其它季节之间的物种数量无显著性差异(P > 0.05)。在空间上,上层水检测到98种,刀鲚、棒花鱼等26种鱼类仅在上层水中检测到。中层水检测到51种,花

根据IUCN红色名录(www.iucnredlist.org)和《动物卷——鱼类》,此次调查的鱼中,极危3种,分别为鳤、云南鲴和长须黄颡鱼;濒危5种,即黑尾
根据生活习性将鱼类划分为5种类型,其中江海洄游型和河海洄游型鱼类各1种,江(河)湖洄游型鱼类25种,河流(山溪)型鱼类10种,湖泊定居型鱼类共51种,占绝对性优势,占比44.74 %。按照鱼类的栖息环境,将其分为中上层(U)、中下层(L)、底层(D)3种类型,中上层鱼类30种;中下层26种;底层38种,占比33.33 % (附表Ⅰ)。
此外,根据鱼类在各样点的总序列丰度大小确定草鱼、鳙、鲤、鲫为本次赣江南昌段调查的优势种[13](附表Ⅱ)。
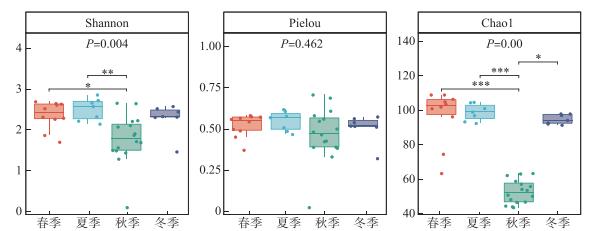
2.3 基于环境DNA宏条形码技术的赣江下游南昌段鱼类α多样性在时间上,不同季节之间的香农指数具有极显著差异(P < 0.01)。夏季和秋季之间具有极显著差异(P < 0.01),春季与秋季之间具有显著性差异(P < 0.05)。Pielou均匀度指数为0.324~0.708,平均值0.504。Pielou均匀度指数在各季节之间无显著性差异(P>0.05)。Chao1指数为43.750~109.000,平均值为79.308,春季和夏季的物种丰富度极显著地高于秋季(P < 0.001),夏季和冬季的物种丰富度显著高于秋季(P < 0.05)(图 3)。从空间上分析得到,香农指数、Pielou均匀度指数、Chao1指数在不同的水层之间没有明显的差异性(P>0.05)(图略)。香农指数、Pielou均匀度指数、Chao1指数在近岸和离岸之间也未显示出明显的差异性(P>0.05)(图略)。
|
图 3 不同季节的多样性指数组间差异 Fig.3 Diversity index differences between groups in different seasons |
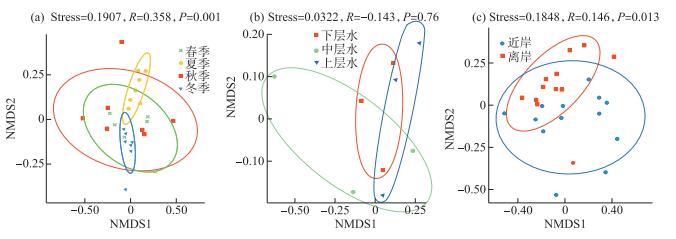
对赣江下游南昌段不同季节、不同水层以及近岸和离岸的鱼类环境DNA信息分别进行了NMDS分析。不同季节的NMDS分析中(图 4a),Stress值为0.1907,ANOSIM分析中,R值大于0,整体P值小于0.01,季节的鱼类环境DNA信息组成具有极显著差异,进一步的ANOSIM分析表明春季和冬季(P=0.904)、秋季与冬季(P=0.434)之间的鱼类环境DNA信息组成无显著差异;春季与夏季(P=0.02)、春季与秋季(P=0.047)之间的环境DNA信息组成具有显著性差异;夏季与秋季(P=0.006)、夏季与冬季(P=0.002)之间的鱼类环境DNA信息组成具有极显著差异。不同水层的NMDS分析中(图 4b),Stress值为0.0322,ANOSIM分析中,R值小于0,整体P值为0.258,说明不同水层组间无显著性差异。进一步的ANOSIM分析表明,上层水与中层水(P=0.209)、上层水与下层水(P=0.379)、中层水与下层水(P=0.300)之间的鱼类环境DNA信息组成均无显著差异。近岸和离岸的NMDS分析中(图 4c),Stress值为0.1848,ANOSIM分析中,R值为0.146,P值小于0.05,说明近岸与离岸之间的鱼类环境DNA信息组成具有显著差异。离岸组与近岸组的差异包含了河流横向上的差异和河流纵向上的差异两个部分。
|
图 4 不同季节、水层、近岸和离岸的NMDS分析图 Fig.4 The NMDS analysis results of different seasons, layers, farshore and inshore |
基于各鱼类物种的序列数进行DCA分析,Lengths of gradient的最大值小于3,因此选择RDA分析进行环境因子关联分析。各环境因子在排序中的相关性系数(r2)以及显著性检验值(P value)见表 1。由表中可得,环境因子中水温(Tem)和pH极显著地影响排序结果(P < 0.001)。RDA分析图中Permutation test值为0.001(附图Ⅰ),即环境因子对鱼类环境DNA信息的影响是极显著的。各鱼类物种与环境因子之间的相关性Heatmap图显示,大部分鱼类的序列相对丰度均与水温呈显著或极显著正相关关系。长薄鳅和须鱊与pH呈显著正相关关系。鲤和圆吻鲴与叶绿素a呈显著正相关关系,鲤与溶解氧呈极显著正相关关系(图 5)。
| 表 1 环境因子对鱼类环境DNA信息的差异性检验 Tab. 1 Difference test of environmental factors on fish environmental DNA information |

|
图 5 鱼类物种与环境因子的相关性Heatmap图 Fig.5 The relevant heatmap between fish species and environmental factors |
本研究基于环境DNA宏条形码技术于2020-2021年调查到的赣江下游南昌段鱼类共114种(附表Ⅰ)。王朝阳[27]在赣江下游南昌样区调查到鱼类41种。敖雪夫[30]在研究水利枢纽对赣江中下游鱼类群落影响时在赣江下游南昌段调查到66种鱼类。苏念等[31]在赣江下游峡江段至南昌段共调查到90种鱼类。王朝阳等[32]对赣江下游一级支流的鱼类多样性进行比较分析,共监测到67种鱼类。王子彤和张鹗[17]基于传统方法研究得出,赣江下游干流调查到鱼类57种。Hu等[28]对赣江下游鱼类的组成、分布以及群落结构进行了调查,在南昌市的样点中检测到鱼类41种,其中33种鱼都在本研究中被检测到。Guo等[33]对赣江中下游鱼类资源的多样性进行调查,在南昌样区检测到鱼类66种,其中与本研究调查到的鱼类重合的有50种。与传统历史数据比较,利用环境DNA宏条形码技术可以检测到更多的物种,尽管也有部分鱼类在传统方法中被检测到而利用环境DNA宏条形码技术未检测到,但是环境DNA宏条形码技术在物种数调查方面显示出的优势体现了该技术在物种监测方面的巨大潜力。
在本研究中,鲤形目鱼类居多,鱼类的生活习性多以湖泊定居型为主,生态型中底层鱼类最多(附表Ⅰ),优势种基本为草鱼、鲤、鲫等鱼类(附表Ⅱ)。在基于传统方法的赣江鱼类的历史研究中[32-33]也是如此,说明基于环境DNA宏条形码技术的鱼类资源检测无论在生态习性、生态型还是优势种方面均与传统方法的调查结果具有一致性,也显示了环境DNA宏条形码技术评估鱼类环境DNA信息组成的有效性及可信性。
本研究基于环境DNA宏条形码技术检测到外来物种,如麦瑞加拉鲮、大口黑鲈、罗非鱼、食蚊鱼(附表Ⅰ),这些外来鱼类在基于传统方法的赣江鱼类资源调查中曾被采集到[17, 28-29]。本研究中还检测到外来物种斑马鱼和美洲鲥,这2种鱼均为养殖鱼类,养殖逃逸或养殖用水流入是检测到这2种鱼的主要原因。在本研究中也检测到了一些海洋鱼类,但是它们的reads数占比均较低,可能是人类喜食鱼,一些海洋鱼类的DNA随着排泄物进入水中而被检测到。此外,本研究基于环境DNA宏条形码技术还检测到了鲤鲫的杂合体,而在传统捕获中并未检测到,这或许是鲤鲫杂合体与鲤或者鲫本身形态差异不大[34],难以区分导致。本研究还在赣江下游南昌段检测到鄱阳湖定居鱼类:纵带鮠、太湖新银鱼、凹尾拟鲿、粘皮鲻鰕虎鱼和间
基于环境DNA宏条形码技术评估赣江下游南昌段鱼类DNA信息的物种多样性和丰富度表明,在不同季节的物种多样性和丰富度方面,春季和夏季检出的鱼类DNA信息的物种多样性显著高于秋季(P < 0.05),春季、夏季和冬季检出的鱼类DNA信息的物种丰富度均显著高于秋季(P < 0.05)(图 3)。在检出的鱼类环境DNA信息的时空变化来看,春季与夏季、春季与秋季、夏季与秋季、夏季与冬季的环境DNA信息组成均具有显著性差异(P < 0.05)(图 4a)。这是因为春末夏初的水体温度适宜鱼类的产卵繁殖[37],鱼类在产卵期间活动也比平时更为活跃,有些鱼类会将精子、卵子直接排入水体中受精,使水体中的环境DNA浓度大大增加,易于检测到更多的物种。这与春季和夏季检测到的鱼类物种DNA信息较多相一致。冬季比秋季检出的鱼类物种数要多这是本研究得出的一个客观的结果,在基于环境DNA宏条形码的鄱阳湖鱼类多样性研究中,鄱阳湖冬季比秋季检出的鱼类物种数也是更多[38]。
在不同水层之间,检出的鱼类DNA信息物种多样性、物种丰富度和鱼类环境DNA信息的时空变化均未显示出显著性差异(P>0.05)(图 4b)。Yamamoto等[39]对日本西部Maizuru海湾的鱼类群落进行了研究,结果表明检测到的上层水的总物种数比下层水更多,物种组成之间存在差异。本研究的结果与Yamamoto等[39]的研究结果(即上、下层水的鱼类物种组成有差异)相矛盾,这是由于河流水体与海洋有不同水文水动力学特征,河流水体通常处于紊流状态,混合程度较大,分层不明显。此外,河流深度较海洋浅,河水温度垂直差异较小,鱼类环境DNA分布不会呈现出垂直分布差异。因此,在较浅的淡水流水系统内进行分层采样是非必要的举措。
在近岸和离岸中,近岸处检测到的物种数量较离岸处多,因为环境DNA会随着水的流动向近岸处汇聚积累[10],也有一些鱼类在繁殖季节会在近岸处产卵,导致环境DNA浓度的增加,如外来物种大口黑鲈每年的5-6月会在近岸处产卵,孵化出的幼鱼也会在近岸处活动[40]。在基于环境DNA宏条形码技术的北京地区3个不同大小的静水系统的鱼类多样性的研究中,近岸处的物种数量大于或等于离岸处[41]。Hayami等[42]也在近岸处检测到更多的物种。本文的研究结果与这些结果一致。
对赣江下游南昌市区域内鱼类的调查表明,不同季节的物种数量多少为之后赣江鱼类资源的调查提供了最好的采样季节的选择。在鱼类的繁殖季节采集水样,可以付出较小努力而检测到更多的物种。季节之间显现的差异性要求基于环境DNA宏条形码技术的调查应该在不同季节进行长时动态监测。另外,因为上层水检测到的物种数量明显多于中层水、下层水,近岸处比离岸处的物种数量更多,因此,对淡水流水系统中的鱼类资源进行调查在近岸处取上层水样即可,除非一些特定鱼类(仅在离岸处检测到的鱼类)的监测。此外,近岸处取样可以避免租船等程序,取样也较为方便。
3.3 赣江下游南昌段所检出鱼类环境DNA信息与环境因子的关系环境因子的变化会对鱼类群落的多样性和群落结构产生影响,从而使鱼类呈现出不同的分布格局[43]。RDA分析结果显示,赣江下游鱼类环境DNA信息组成的主要受水温和pH的影响(表 1,图 5)。因为水温是鱼类群落结构最主要的影响因子,它会影响鱼类的生理、生化和生活史过程[44],对于鱼类性腺的发育和鱼类的产卵繁殖方式起重要作用[45]。䱗、鲫、翘嘴鲌等鱼类未显示出与温度或其他环境因子之间的关系,因为这些鱼类在长江流域有较广的分布,已经适应了该区域的环境的变化,对环境因子的变化具有了一定的耐受性[24]。随着河流水温的升高,水体的溶解氧会出现下降趋势[46],鲤与溶解氧之间为极显著正相关关系,与温度为负相关关系,这一结果与杨少荣等[24]的研究结果一致。
4 附录附表附表Ⅰ、附表Ⅱ和附图Ⅰ见电子版(DOI: 10.18307/2023.0435)。
| 附表Ⅰ 赣江下游南昌段鱼类物种组成 Appendix Ⅰ The fish composition of the Nanchang section of the lower Ganjiang River |
| 附表Ⅱ 基于环境DNA宏条形码技术的赣江南昌段鱼类优势种 Appendix Ⅱ The dominant fish species of the Nanchang section of the lower Ganjiang River based on Environmental DNA metabarcoding |

|
附图Ⅰ 鱼类物种与环境因子的冗余分析 AttachedFig1 The RDA analysis between fish community and environmental factors |
致谢: 感谢陈金萍、雷姚、赵帅、黄建华在水样采集过程中提供的支持。感谢李雄辉、黎敏教授、王文娟副研究员在数据分析中给予的帮助。
| [1] |
Strayer DL, Dudgeon D. Freshwater biodiversity conservation: Recent progress and future challenges. Journal of the North American Benthological Society, 2010, 29(1): 344-358. DOI:10.1899/08-171.1 |
| [2] |
Collen B, Whitton F, Dyer EE et al. Global patterns of freshwater species diversity, threat and endemism. Global Ecology and Biogeography, 2014, 23(1): 40-51. DOI:10.1111/geb.12096 |
| [3] |
Zhou S, Fan CR, Xia HM et al. Combined use of eDNA metabarcoding and bottom trawling for the assessment of fish biodiversity in the Zhoushan Sea. Frontiers in Marine Science, 2022, 8: 809703. DOI:10.3389/fmars.2021.809703 |
| [4] |
Zou KS, Chen JW, Ruan HT et al. eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling. Science of the Total Environment, 2020, 702: 134704. DOI:10.1016/j.scitotenv.2019.134704 |
| [5] |
Oka SI, Doi H, Miyamoto K et al. Environmental DNA metabarcoding for biodiversity monitoring of a highly diverse tropical fish community in a coral reef lagoon: Estimation of species richness and detection of habitat segregation. Environmental DNA, 2021, 3(1): 55-69. DOI:10.1002/edn3.132 |
| [6] |
Thomsen PF, Willerslev E. Environmental DNA—An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation, 2015, 183: 4-18. DOI:10.1016/j.biocon.2014.11.019 |
| [7] |
Cristescu ME, Hebert PDN. Uses and misuses of environmental DNA in biodiversity science and conservation. Annual Review of Ecology, Evolution, and Systematics, 2018, 49: 209-230. DOI:10.1146/annurev-ecolsys-110617-062306 |
| [8] |
Deiner K, Bik HM, Mächler E et al. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology, 2017, 26(21): 5872-5895. DOI:10.1111/mec.14350 |
| [9] |
Evans NT, Olds BP, Renshaw MA et al. Quantification of mesocosm fish and amphibian species diversity via environmental DNA metabarcoding. Molecular Ecology Resources, 2016, 16(1): 29-41. DOI:10.1111/1755-0998.12433 |
| [10] |
Hänfling B, Lawson Handley L, Read DS et al. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods. Molecular Ecology, 2016, 25(13): 3101-3119. DOI:10.1111/mec.13660 |
| [11] |
Valentini A, Taberlet P, Miaud C et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [12] |
Wang M, Yang X, Wang W et al. Fish diversity in Chongqing section of the national nature reserve for rare and endemic fish in the Upper Yangtze River based on edna technology. Acta Hydrobiologica Sinica, 2022, 46(1): 2-16. [王梦, 杨鑫, 王维等. 基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究. 水生生物学报, 2022, 46(1): 2-16. DOI:10.7541/2021.2021.132] |
| [13] |
Shu L, Lin JY, Xu Y et al. Investigating the fish diversity in Erhai Lake based on environmental DNA metabarcoding. Acta Hydrobiologica Sinica, 2020, 44(5): 1080-1086. [舒璐, 林佳艳, 徐源等. 基于环境DNA宏条形码的洱海鱼类多样性研究. 水生生物学报, 2020, 44(5): 1080-1086.] |
| [14] |
王杰, 王保畲, 罗正齐. 长江大辞典. 武汉: 武汉出版社, 1997.
|
| [15] |
《中国河湖大典》编纂委员会. 中国河湖大典. 北京: 中国水利水电出版社, 2010.
|
| [16] |
Tong LL, Guo CB, Wang R et al. Research status of water environment and fish resources under multiple human disturbances in the Gan-Jiang River Basin: A review. Resources and Environment in the Yangtze Basin, 2019, 28(12): 2879-2892. [仝路路, 郭传波, 王瑞等. 多重人类活动干扰下赣江流域水环境和鱼类资源的研究现状分析. 长江流域资源与环境, 2019, 28(12): 2879-2892. DOI:10.11870/cjlyzyyhj20191209] |
| [17] |
Wang ZT, Zhang G. An updated species checklist of freshwater fishes from the Ganjiang River. Biodiversity Science, 2021, 29(9): 1256-1264. [王子彤, 张鹗. 赣江鱼类物种更新名录. 生物多样性, 2021, 29(9): 1256-1264. DOI:10.17520/biods.2021119] |
| [18] |
Littlefair JE, Hrenchuk LE, Blanchfield PJ et al. Thermal stratification and fish thermal preference explain vertical eDNA distributions in lakes. Molecular Ecology, 2021, 30(13): 3083-3096. DOI:10.1111/mec.15623 |
| [19] |
Li JL, Hatton-Ellis TW, Lawson Handley LJ et al. Ground-truthing of a fish-based environmental DNA metabarcoding method for assessing the quality of lakes. Journal of Applied Ecology, 2019, 56(5): 1232-1244. DOI:10.1111/1365-2664.13352 |
| [20] |
Taberlet P, Bonin A, Zinger L et al. Environmental DNA: For biodiversity research and monitoring. Oxford University Press, 2018.
|
| [21] |
陈宜瑜. 中国动物志-硬骨鱼纲-鲤形目中卷. 北京: 科学出版社, 1998.
|
| [22] |
乐佩奇. 中国动物志-硬骨鱼纲-鲤形目下卷. 北京: 科学出版社, 2000.
|
| [23] |
朱松泉. 中国淡水鱼类检索. 南京: 江苏科学技术出版社, 1995.
|
| [24] |
Yang SR, Li MZ, Zhu QG et al. Spatial and temporal variations of fish assemblages in Poyanghu Lake. Resources and Environment in the Yangtze Basin, 2015, 24(1): 54-64. [杨少荣, 黎明政, 朱其广等. 鄱阳湖鱼类群落结构及其时空动态. 长江流域资源与环境, 2015, 24(1): 54-64. DOI:10.11870/cjlyzyyhj201501008] |
| [25] |
Xiong GY. Preliminary investigation of fish resources in internal lakes of Poyang Lake wetland. Jiangsu Agricultural Sciences, 2018, 46(21): 186-190. [熊国勇. 鄱阳湖区域湿地内湖鱼类资源初步调查. 江苏农业科学, 2018, 46(21): 186-190. DOI:10.15889/j.issn.1002-1302.2018.21.048] |
| [26] |
Zhu ZB, Wang YP, Lin DQ et al. Species diversity of fish in Jingjiang section near the coast of the Yangtze River Estuary. Journal of Anhui Agricultural University, 2021, 48(4): 614-619. [朱宗波, 王银平, 蔺丹清等. 长江近口靖江段近岸鱼类群落多样性. 安徽农业大学学报, 2021, 48(4): 614-619.] |
| [27] |
王朝阳. 赣江下游鱼类群落结构及物种多样性的分析[学位论文]. 南昌: 南昌大学, 2019.
|
| [28] |
Hu ML, Wang CY, Liu YZ et al. Fish species composition, distribution and community structure in the lower reaches of Ganjiang River, Jiangxi, China. Scientific Reports, 2019, 9: 10100. DOI:10.1038/s41598-019-46600-2 |
| [29] |
Shi ZM, Zhang JM, Wu HJ et al. Taxonomic diversity pattern and composition of fish species in the upper reaches of Ganjiang River, Jiangxi, China. PLoS One, 2020, 15(11). DOI:10.1371/journal.pone.0241762 |
| [30] |
敖雪夫. 水利枢纽对赣江中下游鱼类群落结构影响[学位论文]. 南昌: 南昌大学, 2016.
|
| [31] |
Su N, Li L, Xu ZQ et al. Status of fish resources in Xiajiang to Nanchang section of Ganjiang River. Journal of Huazhong Agricultural University, 2012, 31(6): 756-764. [苏念, 李莉, 徐哲奇等. 赣江峡江至南昌段鱼类资源现状. 华中农业大学学报, 2012, 31(6): 756-764.] |
| [32] |
Wang CY, Huang DC, Hu ML et al. Comparative analysis of fish community diversity in two primary tributaries of the lower Ganjiang River. Journal of Hydroecology, 2019, 40(3): 99-106. [王朝阳, 黄德才, 胡茂林等. 赣江下游一级支流鱼类多样性比较分析. 水生态学杂志, 2019, 40(3): 99-106. DOI:10.15928/j.1674-3075.2019.03.014] |
| [33] |
Guo Q, Liu XJ, Ao XF et al. Fish diversity in the middle and lower reaches of the Ganjiang River of China: Threats and conservation. PLoS One, 2018, 13(11): e0205116. DOI:10.1371/journal.pone.0205116 |
| [34] |
Zhu SL, Bai QL, Cao DC et al. Comparison of morphological characteristics of the same parent F1 hybrid between Alpine carp and Crucian carp. Northern Chinese Fisheries, 1999(4): 19-20. [朱世龙, 白庆利, 曹顶臣等. 高寒鲤、红鲫杂交F-1同亲本形态学特征比较. 黑龙江水产, 1999(4): 19-20.] |
| [35] |
傅萃长. 长江流域鱼类多样性空间格局与资源分析-兼论银鱼的生物多样性与系统发育[学位论文]. 上海: 复旦大学, 2003.
|
| [36] |
Zhang X, Yang TY, Luo XH et al. Fish phylogenetic community structure in the Poyang Lake and its tributary the Xiushui River in summer. Acta Hydrobiologica Sinica, 2020, 44(6): 1297-1312. [张旭, 杨婷越, 罗小红等. 鄱阳湖湖区及支流修水夏季鱼类系统发育群落结构分析. 水生生物学报, 2020, 44(6): 1297-1312. DOI:10.7541/2020.151] |
| [37] |
Duan XB, Chen DQ, Li ZH et al. Current status of spawning grounds of fishes with pelagic eggs in the middle reaches of the Yangtze River after impoundment of the Three Gorges Reservior. Journal of Fishery Sciences of China, 2008, 15(4): 523-532. [段辛斌, 陈大庆, 李志华等. 三峡水库蓄水后长江中游产漂流性卵鱼类产卵场现状. 中国水产科学, 2008, 15(4): 523-532.] |
| [38] |
陈金萍. 基于环境DNA宏条形码的鄱阳湖蚌类和鱼类多样性研究[学位论文]. 南昌: 南昌大学, 2021.
|
| [39] |
Yamamoto S, Masuda R, Sato Y et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea. Scientific Reports, 2017, 7: 40368. DOI:10.1038/srep40368 |
| [40] |
Nakai K. Black bass nadonogairaigyoniyoru seitaitekieikyou. Yousui to Haisui, 2004, 46: 48-56. |
| [41] |
Zhang S, Lu Q, Wang YY et al. Assessment of fish communities using environmental DNA: Effect of spatial sampling design in lentic systems of different sizes. Molecular Ecology Resources, 2020, 20(1): 242-255. DOI:10.1111/1755-0998.13105 |
| [42] |
Hayami K, Sakata MK, Inagawa T et al. Effects of sampling seasons and locations on fish environmental DNA metabarcoding in dam reservoirs. Ecology and Evolution, 2020, 10(12): 5354-5367. DOI:10.1002/ece3.6279 |
| [43] |
Gao TY, Xie D, Peng ND et al. Fish community structure and its relationships with environmental factors in the Beijiang River. Freshwater Fisheries, 2017, 47(6): 47-55. [高天扬, 谢迪, 彭宁东等. 北江鱼类群落结构及其与环境因子的关系. 淡水渔业, 2017, 47(6): 47-55.] |
| [44] |
Wang LM, Hu HJ, Wang D. Ecological impacts of disconnection from the Yangtze on fish resources in Zhangdu Lake. Resources and Environment in the Yangtze Basin, 2005, 14(3): 287-292. [王利民, 胡慧建, 王丁. 江湖阻隔对涨渡湖区鱼类资源的生态影响. 长江流域资源与环境, 2005, 14(3): 287-292. DOI:10.3969/j.issn.1004-8227.2005.03.005] |
| [45] |
殷名称. 鱼类生态学. 北京: 中国农业出版社, 1993.
|
| [46] |
Shen C, Lv PY, Feng SX et al. Study on the effect of the Xiangjiaba Reservoir's impoundment on the dissolved oxygen saturation in the downstream river. Freshwater Fisheries, 2014, 44(6): 31-36. [沈忱, 吕平毓, 冯顺新等. 向家坝水库蓄水对下游江段溶解氧饱和度影响研究. 淡水渔业, 2014, 44(6): 31-36.] |
 2023, Vol. 35
2023, Vol. 35 

