生物多样性是人类生存和发展的基础,保护生物多样性有助于维护地球家园,促进人类可持续发展。水生无脊椎动物是水生生物多样性的重要组成部分,主要包括轮虫、环节动物、线虫动物、节肢动物和软体动物等,在水生生态系统物质循环、能量流动和信息传递中起着关键作用[1-2]。水生无脊椎动物对水质变化敏感,可作为指示生物反映气候变化和水质污染[3-4]。因此,研究水生无脊椎动物及其群落结构组成,对了解水生生物多样性、水质评价和保护水体健康状态具有重要意义。
对于无脊椎动物调查,目前仍以形态学鉴定为主,但形态学鉴定对技术人员的要求高、容易鉴定错误[5],且消耗成本较高[6]。环境DNA宏条形码(environmental DNA metabarcoding, eDNA)技术是指从环境中提取DNA,随后利用高通量测序扩增,以获得大量DNA序列,再通过与数据库中已知序列比对,得到环境中物种信息的过程[7-9]。因其成本低、操作方便、灵敏度高等特点[6],常被应用于水生生物调查中。在检测无脊椎动物物种组成方面更为快速、准确和全面[10],能对稀有种和入侵物种进行有效监测[11-12]。研究表明,基于eDNA的分子技术较为可靠[13],并且已成为生物多样性评估的重要手段[9, 14]。
海珠湖位于广东省广州市,与部分河涌共同组成了广州海珠国家湿地公园,其具有独特的供给、调节气候和文化旅游功能,对华南动植物的生物多样性保护具有重要作用。目前,关于海珠湖生物多样性和水质研究,主要集中在湿地植物、重金属、浮游植物和鸟类等方面[15],海珠湖水生无脊椎动物的调查资料较少,偶有涉及[16]。为全面了解海珠湖的生物多样性,利用eDNA技术分析海珠湖的水生无脊椎动物群落结构,并结合传统形态学调查方法鉴定浮游动物,为广州海珠国家湿地公园海珠湖水生生态监测体系和生物多样性保护提供基础资料和科学依据。
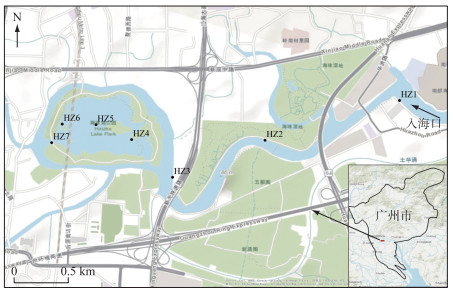
1 材料与方法 1.1 采样点位海珠湖位于广州市海珠区,经多条河涌与珠江相连,是广州新中轴上的生态焦点。根据湖泊形态和水动力学特征,于2021年1月对海珠湖7个站点进行生态调查(图 1)。HZ1~HZ3位于与海珠湖连接的最大的一条河涌——石榴岗河,HZ1位于公园外的第一个拐点处,且靠近工厂及居民住宅区,HZ2位于公园内果树林旁,HZ3为海珠湖与石榴岗河的交汇处,HZ4~HZ7位于海珠湖湖区,HZ4位于湖区东侧的游船码头,HZ5位于湖区中央,HZ6位于湖区西侧的观鸟亭,HZ7为湖区西侧的泄洪口(水闸)处。7个采样点分布较为分散,且较有代表性,能够反映海珠湖的水体状况。
|
图 1 海珠湖采样点分布 Fig.1 Distribution of sampling sites in Lake Haizhu |
用YSI plus水质分析仪(美国)对水温、pH值、溶解氧(DO)和电导率(EC)进行现场测定;使用塞氏盘测定水体透明度(SD)。使用采水器采集水样带回实验室,按照《水和废水监测分析方法》[17]测定总氮(TN)、氨氮(NH3)、硝酸盐(NO3-)、亚硝酸盐(NO2-)、总磷(TP)和磷酸盐(PO43-)浓度,用丙酮分光光度法测定叶绿素a(Chl.a)浓度。
1.3 浮游动物形态学样品采集定性样品:使用孔径64 μm浮游生物网水平和垂直拖拽采集。浮游甲壳动物定量样品:使用64 μm孔径浮游生物网过滤浓缩20 L水样;轮虫定量样品:使用20 μm孔径浮游生物网过滤浓缩5 L水样。样品均收集至50 mL聚乙烯瓶中,加入甲醛溶液固定保存,带回实验室镜检和计数。
1.4 3种eDNA样品的采集与处理水采样品(W):用5 L有机玻璃采水器采集表层水体样品,取1 L水样进行抽滤。网采样品(N):使用64 μm浮游生物网拖拽,收集样品后抽滤。沉积物样品(S):使用采泥器对海珠湖表层沉积物进行采集,称量80 g沉积物样品进行蔗糖浮选[18],对浮选后的样品进行抽滤,获得休眠卵样品。
样品均使用孔径0.22 μm、直径50 mm的滤膜抽滤,抽滤后立即进行DNA提取,并在-80℃进行保存。沉积物样品的处理中,使用蔗糖浮选提取出沉积物中的休眠卵,如轮虫、桡足类、线虫等均具有休眠卵,但不排除提取的样品中存在无脊椎动物的残体或分泌物等情况。
1.5 DNA提取、PCR扩增和高通量测序共采集了6个水采样品(W1~W6)、7个网采样品(N1~N7)和4个沉积物样品(S1、S3~S5)。采用DNeasy Power土壤试剂盒(QIAGEN12888, Germany)从样品中提取DNA,完成后用NanoDrop对DNA进行质量评价。使用真核生物通用引物528F(5’-GCGGTAATTCCAGCTCCAA-3’)和760R(5’-AATCCRAGAATTTCACCTCT-3’)对18S rRNA基因V4区进行扩增[19]。PCR的反应体系(共20 μL)为:4 μL 5×FastPfu Buffer、2 μL 2.5 mmol/L dNTPs、0.8 μL Forward Primer(5 μmol/L)、0.8 μL Reverse Primer(5 μmol/L)、0.4 μL FastPfu Polymerase、0.2 μL BSA、10 ng Template DNA,补ddH2O至20 μL。用ABI GeneAmpⓇ 9700型PCR仪进行扩增,反应程序为:于95℃条件下变性3 min,循环30次(95℃变性30 s,60℃退火30 s,72℃延伸45 s),最后72℃延伸10 min。PCR产物用2 % 琼脂糖凝胶电泳检测,完成检测并回收后,参照电泳初步定量结果用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行定量检测。纯化后的产物在Illumina MiSeq平台(Illumina, San Diego, USA)上进行测序。由上海美吉生物医药科技有限公司提供测序服务。
1.6 数据统计分析Miseq测序得到的PE reads首先根据overlap关系进行拼接,同时对序列质量进行质控和过滤。区分样本后使用Uparse(http://www.drive5.com/uparse/)按照97 % 相似性对非重复序列(不含单序列)进行可操作分类单元(operational taxonomic unit, OTU)聚类,在聚类过程中去除嵌合体,得到OTU的代表序列。为得到每个OTU对应的物种分类信息,使用RDP classifier贝叶斯算法(https://sourceforge.net/projects/rdp-classifier/)比对到美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)中的核酸序列数据库(ftp://ftp.ncbi.nih.gov/blast/db/)进行物种注释。使用mothur计算Sobs指数,利用Networkx完成网络图绘制,使用Canoco 4.5完成RDA分析,利用R语言工具及Graphpad Prism 9.0完成其余图形绘制。
2 结果 2.1 测序结果对海珠湖的17个样品进行高通量测序,共获得1036384条优化序列,平均长度为306 bp。为保证数据可比性,按照最小样本序列数对测序结果进行随机抽平处理,17个样品最终得到679320条序列(占抽平前生物序列的65.55 %)。将抽平后的数据按照97 % 的相似水平进行聚类,获得1130个OTUs。去除细菌、真菌和浮游植物等,最终获得137个无脊椎动物OTUs。所有后续分析均基于此数据。
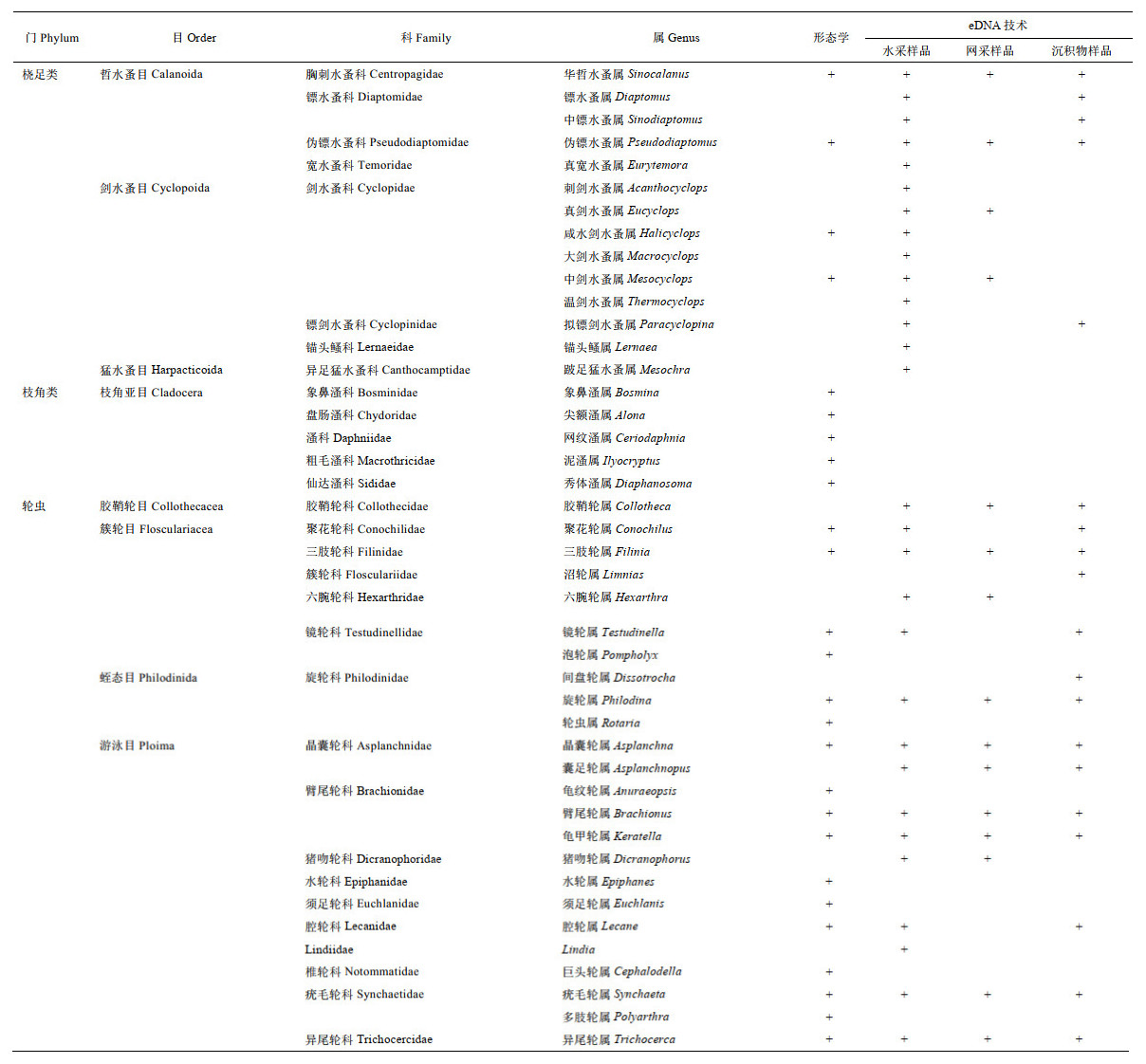
在无脊椎动物序列中,轮虫动物门最为丰富,序列数占总序列数的44.48 %,其次是环节动物门与节肢动物门,分别占25.85 % 和24.66 %,随后是线虫动物门(3.09 %)、扁形动物门(0.71 %)、外肛动物门(0.27 %)、软体动物门(0.11 %)、腹毛动物门(0.03 %)和缓步动物门(0.01 %)。依据NCBI-NT数据库中BLAST的最佳值,对eDNA测序结果进行注释,137个无脊椎动物OTUs分属于9门16纲34目71科93属(附表Ⅰ),其中,环节动物门、节肢动物门、外肛动物门、腹毛动物门、软体动物门、线虫动物门、扁形动物门、轮虫动物门、缓步动物门分别检出11、42、3、3、5、40、5、23、2个OTUs,还有3个无脊椎动物OTUs未注释到门。
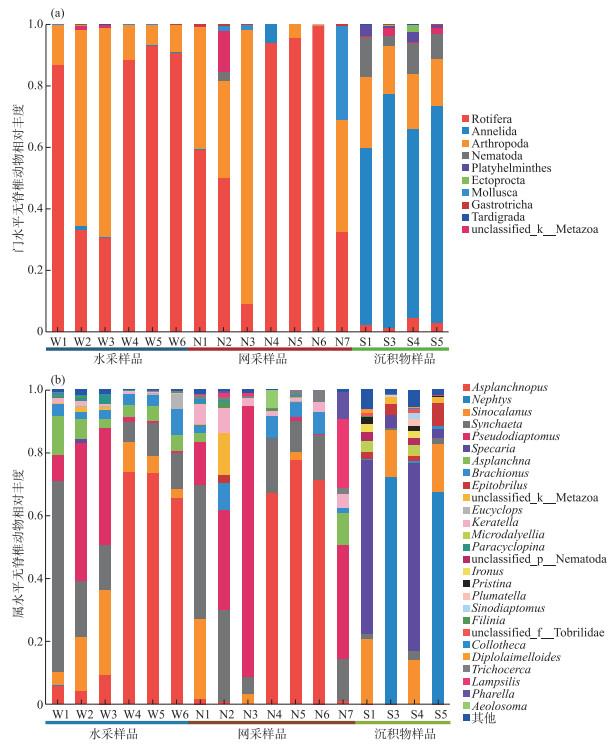
2.2 海珠湖无脊椎动物群落组成 2.2.1 水采样品、网采样品和沉积物中无脊椎动物的群落组成海珠湖水采样品和网采样品共检出无脊椎动物9门13纲22目41科52属70个OTUs。其中,水采样品共检出9门13纲21目39科50属68个OTUs,轮虫动物门和节肢动物门为水采样品中的主要门类(图 2a),平均相对丰度分别为70.47 % 和28.67 %。属水平上,囊足轮属(Asplanchnopus)、疣毛轮属(Synchaeta)、伪镖水蚤属(Pseudodiaptomus)和华哲水蚤属(Sinocalanus)是水采样品的主要组成部分,其平均相对丰度分别为38.74 %、20.23 %、15.38 % 和11.04 % (图 2b)。
|
图 2 海珠湖门(a)和属(b)水平无脊椎动物相对丰度组成 Fig.2 Relative abundance of invertebrates on phylum (a) and genus (b) level in Lake Haizhu |
网采样品共检出无脊椎动物6门9纲13目22科27属35个OTUs,OTU数约为水采样品的50 %,主要门类为轮虫动物门和节肢动物门,平均相对丰度分别为62.87 % 和28.78 % (图 2a)。属水平上,囊足轮属、伪镖水蚤属、疣毛轮属和华哲水蚤属是网采样品的主要组成部分,其平均相对丰度分别为31.35 %、24.18 %、18.90 % 和4.52 % (图 2b)。
沉积物无脊椎动物休眠卵OTU丰富度比水体高,共检出9门13纲26目53科70属103个OTUs,主要门类为环节动物门和节肢动物门,平均相对丰度分别为66.54 % 和17.85 % (图 2a)。环节动物门在S3和S5中有较高的相对丰度,分别为76.25 % 和70.65 %,而在S1和S4相对丰度较低;而节肢动物门在S1和S4有较高的相对丰度,在S3和S5中相对丰度较低。沉积物样品中的无脊椎动物优势属为齿吻沙蚕属(Nephtys)、斯必卡虫属(Specaria)和华哲水蚤属,分别占34.96 %、30.59 % 和16.24 % (图 2b)。齿吻沙蚕属在S3和S5中有更高的相对丰度,分别为72.28 % 和67.54 %;在S1和S4相对丰度较低,均低于1 %。斯必卡虫属在S1和S4中有更高的相对丰度,分别为55.43 % 和59.87 %;在S3和S5相对丰度较低,均低于4 %。而华哲水蚤属的丰度在各个站点较为一致,相对丰度范围为14 % ~21 %。
水采样品和网采样品的OTU组成相似性较高,但也存在差异(图 2)。环节动物门、节肢动物门、外肛动物门、腹毛动物门、软体动物门、线虫动物门、扁形动物门、轮虫动物门和缓步动物门在水采样品中均被检出,但在网采样品中未检出外肛动物门、扁形动物门和缓步动物门。水采样品的OTU数高于网采样品,两者的丰度组成也不相同(图 2)。两种类型样品中,节肢动物门和轮虫动物门均为水体样品中的主要门类,但水采样品中轮虫动物门相对丰度比网采样品高7.61 %。超过50 % 点位的轮虫动物门相对丰度在86 % 以上,但点位HZ2和HZ3的轮虫动物门丰度较低,在样品N2、N3和W3中节肢动物门成为第一优势门类,相对丰度分别为63.86 %、67.97 % 和89.44 %。就属水平而言,囊足轮属、伪镖水蚤属、疣毛轮属和华哲水蚤属是水体样品中主要类群,分别占水采样品和网采样品的85.39 % 和78.95 % (图 3)。两种水体样品中,轮虫动物门的囊足轮属均为第一优势属,点位HZ4、HZ5和HZ6的囊足轮属相对丰度均在65 % 以上,而点位HZ1、HZ2和HZ3其相对丰度均在10 % 以下,可能与HZ1~HZ3站点位于河涌,HZ4~HZ6位于湖区有关。轮虫动物门的疣毛轮属是水采样品的第二大属,但在网采样品中节肢动物门的伪镖水蚤属是第二大属。伪镖水蚤属在点位HZ2和HZ3有较高丰度,且成为W2、W3、N2和N3的第一优势属。疣毛轮属在点位HZ1有较高丰度,且成为W1和N1的第一优势属。

|
图 3 无脊椎动物的属丰富度:(a)所有样品(共93属);(b)水采样品(共50属);(c)网采样品(共27属);(d)沉积物样品(共70属) Fig.3 Genus richness of invertebrates: (a) all samples (93 genera); (b) samples directly collected from water (50 genera); (c) samples collected by a planktonic net (27 genera); (d) sediment samples (70 genera) |
海珠湖水体和沉积物样品中共检出无脊椎动物9门93属,其中,沉积物样品中有70属(类),丰富度最高,占所有无脊椎动物属水平丰富度的75.27 %,其次为水采样品,共检出50属,占53.76 %,网采所获得的属类最少,为27属,占29.03 % (图 3)。在所有样品中,线虫动物门的属最多,共29属,其次是节肢动物门和轮虫动物门,分别为27属和17属,与沉积物样品的属分布较为接近(图 3d)。水采样品中节肢动物门和轮虫动物门的属最多,均为15属(图 3b);网采样品中轮虫动物门的属最多,为11属,环节动物门、软体动物门和节肢动物门次之,均有4属(图 3c)。水体网采样品中未检出外肛动物门、扁形动物门和缓步动物门,而这3门在水采样品和沉积物样品中均检出5属,其相对丰度较低,在水采样品和沉积物样品中3个门合计分别占2.98 % 和0.01 %。
2.2.2 水采样品、网采样品和沉积物中无脊椎动物的群落组成差异非度量多维尺度分析(non-metric multidimensional scaling, NMDS)揭示了无脊椎动物群落具有明显的时空分化特征。其压力值为0.06(< 0.2)表明结果可信。由图 4a可知,水采、网采和沉积物样本分布在不同位置,无脊椎动物组成存在显著性差异(P≤0.05)。Sobs指数为观测到的OTU数,沉积物样品Sobs指数(67±13.39)最高,水采样品(30.67±10.41)次之,网采样品(16±6.08)最低,且3种样品之间存在显著差异(P≤0.05)(图 4b)。

|
图 4 水采样品、网采样品和沉积物样品的NMDS(a)及Sobs指数均值差异(b) (*、**和***分别代表显著水平P < 0.05、P < 0.01、P < 0.001, 下同) Fig.4 NMDS (a) and comparison of mean values of Sobs index (b) of three samples (** and *** represent significant levels P < 0.05, P < 0.01, and P < 0.001, respectively) |
网络分析能够揭示无脊椎动物群落中的物种相互作用,以及群落中占有重要地位的类群。根据OTU在所有样品中的丰度组成,选择总丰度前50 % 的属进行Spearman相关分析,再筛选相关系数绝对值大于或等于0.5且P值小于0.05的数值构建相关性网络图,从而确定海珠湖无脊椎动物中的重要类群以及无脊椎动物优势类群之间的相互作用。
水采样品无脊椎动物网络图中包含45个节点,147条边;网采样品无脊椎动物网络图中包含25个节点,48条边;沉积物休眠卵的无脊椎动物网络图中包含44个节点,202条边(图 5)。三者相比,沉积物休眠卵样品具有更高的平均度(average degree),其次为水采样品,网采样品最低。水采样品、网采样品和沉积物休眠卵样品正相关的边数分别为119条、45条和193条,正相关关系均超80 %,表明海珠湖无脊椎动物群落结构较为稳定,且物种间倾向于协同互作。

|
图 5 无脊椎动物属水平的相关性网络图(a:水采样品;b:网采样品;c:沉积物样品) (图中节点的大小表示物种丰度,不同颜色表示不同的物种;连线的颜色表示正负相关性,红色表示正相关,绿色表示负相关;线的粗细表示相关性系数的大小,线越粗,表示物种之间的相关性越高;线越多,表示该物种与其他物种之间的联系越密切) Fig.5 Network analysis of the invertebrate community at genus level (a: samples directly collected from water; b: samples collected by a planktonic net; c: sediment samples) (The size of the nodes within graph indicates species abundance, and different colors indicate different species; the color of the connecting lines indicates positive and negative correlation, red indicates positive correlation and green indicates negative correlation; the thickness of the lines indicates the size of the correlation coefficient, the thicker the line, the higher the correlation between species; the more lines, the closer the connection between the species and other species) |
水采样品中,拟镖剑水蚤属(Paracyclopina)的介数中心性(0.06)最高,它的节点为关键节点,同时拟镖剑水蚤属也是水采样品无脊椎动物在维持群落结构稳定起到重要作用的关键物种。网采样品中,伪镖水蚤属的介数中心性(0.18)最高,为水采样品无脊椎动物的关键物种。沉积物休眠卵样品中,所有节点的介数中心性均为0,网络中没有观察到网络关键节点。
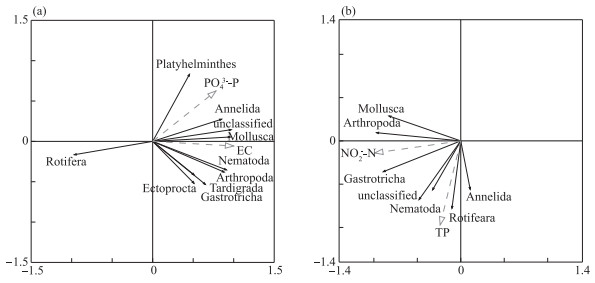
2.4 水体样品群落结构与环境因子的关系 2.4.1 门水平群落结构与环境因子的关系以不同海珠湖无脊椎动物门水平群落丰度为响应变量,环境因子为解释变量进行分析。水采和网采样品进行无脊椎动物门水平除趋势对应分析(detrended correspondence analysis, DCA),排序结果显示4个轴的最大长度分别为0.86和1.86,因此使用冗余分析(redundancy analysis, RDA)。经蒙特卡洛置换检验和前向选择后,各保留了2个对门水平无脊椎动物群落结构产生显著影响的环境因子(P < 0.05)(图 6)。RDA排序轴的全部特征值分别解释了水采和网采无脊椎动物群落变异程度的98.4 % 和96.3 %,前两轴分别解释了变异程度的89.2 % 和75.8 % (附表Ⅱ和附表Ⅲ),基本反映了海珠湖无脊椎动物与环境因子之间的生态关系。
|
图 6 环境因子与海珠湖无脊椎动物门水平的冗余分析排序图(a:水采样品;b:网采样品) Fig.6 Redundancy analysis between invertebrates on phylum level and environmental factors in Lake Haizhu (a: samples directly collected from water; b: samples collected by a planktonic net) |
RDA分析显示,水采样品中,磷酸盐(P < 0.05)和电导率(P < 0.01)是影响无脊椎动物群落结构的主要环境因子,且都与第一轴呈正相关。轮虫动物门与磷酸盐和电导率呈负相关关系;其余无脊椎动物均与磷酸盐和电导率呈正相关关系(图 6a)。网采样品中,亚硝酸盐(P < 0.01)和总磷(P < 0.05)是影响无脊椎动物群落结构的主要环境因子,亚硝酸盐和总磷与无脊椎动物均呈正相关(图 6b)。RDA分析表明,电导率和亚硝酸盐分别是影响水采和网采无脊椎动物最重要的环境因子。
2.4.2 属水平群落结构与环境因子的关系为进一步揭示影响海珠湖水体无脊椎动物群落组成的环境因子,对分类水平总丰度前30 % 的属与显著相关的两个环境因子进行Spearman相关性分析。在水采样品中(图 7a),电导率和磷酸盐与大部分无脊椎动物正相关,如伪镖水蚤属、拟沼螺属和龟甲轮属(Keratella)等;少部分的无脊椎动物与之负相关,如中剑水蚤属(Mesocyclops)、中镖水蚤属(Sinodiaptomus)和异尾轮属(Trichocerca)等。在网采样品中(图 7b),除囊足轮属、真剑水蚤属(Eucyclops)和顠体虫属(Aeolosoma)等5属,大部分无脊椎动物均与亚硝酸盐和总磷正相关。
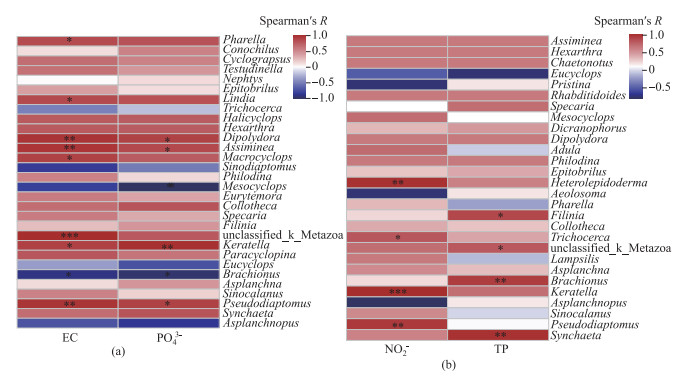
|
图 7 环境因子与海珠湖无脊椎动物属水平相关性热图(a:水采样品;b:网采样品) Fig.7 Correlation heatmap between environmental factors and invertebrates on genus levels in Lake Haizhu (a: samples directly collected from water; b: samples collected by a planktonic net) |
基于eDNA技术共检测到21个轮虫OTU和26个桡足类OTU,注释出桡足类14属和轮虫17属,没有注释到枝角类的OTU(附表Ⅳ)。为验证eDNA技术的可靠性,同步对轮虫动物门、节肢动物门甲壳纲的桡足类和枝角类进行形态学鉴定。
在本次海珠湖调查的7个站点中,使用形态学方法共鉴定出浮游动物26属,水采样品的eDNA共注释出浮游动物29属,网采样品的eDNA共注释出15属,沉积物休眠卵的eDNA共注释出19属(图 8)。4种方法共同发现的浮游动物为9属,包含轮虫7属和桡足类2属(附表Ⅳ和图 8)。仅在形态学中鉴定出的浮游动物共12属,其中包含枝角类5属和轮虫7属;仅在eDNA中检测出的浮游动物有17属,包括桡足类10属和轮虫7属。

|
图 8 使用eDNA技术和形态学鉴定属水平的浮游动物丰富度(a:浮游动物属丰富度的韦恩图;b:桡足类属丰富度的韦恩图;c:轮虫属丰富度的韦恩图;d:浮游动物属丰富度的比较) Fig.8 Identification of zooplankton on genus level usinge DNA and morphology (a: venn diagram of zooplankton genus richness; b: venn diagram of copepod genus richness; c: venn diagram of rotifer genus richness; d: comparison of zooplankton genus richness) |
形态学样品中鉴定轮虫17属,桡足类4属和枝角类5属,其中臂尾轮属(Brachionus)、多肢轮属(Polyarthra)和龟甲轮属为主要组成部分(图 9),其平均丰度分别为(38±35)、(30±22)和(39±60)ind./L,三者的总丰度占形态学鉴定所有浮游动物的64.14 %;臂尾轮属、多肢轮属和龟甲轮属丰度最高值均出现在HZ1,分别为123、81和183 ind./L。

|
图 9 海珠湖浮游动物属水平相对丰度(HZ1~HZ7表示点位,W为水采样品测序结果,N为网采样品测序结果,S为沉积物休眠卵样品测序结果,M为形态学样品鉴定结果) Fig.9 Percentage of zooplankton abundance on genus level in Lake Haizhu (HZ1-HZ7 means sampling sites; W means samples directly collected from the water, N means samples collected by a planktonic net, S means sediment samples, M means samples identified based on the morphology) |
eDNA技术采样方便、检测速度快,具有检测出形态学难以鉴定和稀少物种的潜力[9, 14, 20],已成为生态调查的重要工具,在水生生物调查中已有较多应用[21-22]。例如,对丹麦的5条溪流无脊椎动物进行调查,发现eDNA技术与形态学鉴定的结果有重叠的部分,又可以相互补充[7];eDNA的检测结果与实际物种组成相符[23],是记录河流无脊椎动物的可靠工具[13];太湖的底栖动物监测结果表明eDNA技术可以检测到更多的底栖动物类群,且与形态学方法的鉴定结果具有相似性[10]。
本次利用eDNA注释出的海珠湖无脊椎动物种类在以往周边水域调查中大部分都有记录。2020年对海珠湿地大型底栖动物的调查中共检出环节动物门13属,其中包含齿吻沙蚕属和顠体虫属[16],本研究共检出环节动物门5属,分别为顠体虫属、吻盲虫属(Pristina)、斯必卡虫属、Dipolydora属和齿吻沙蚕属,其中齿吻沙蚕属相对丰度最高。2015年对珠江三角洲河网调查中发现桡足类24属,其中常见优势属有中剑水蚤属和华哲水蚤属等[24],本次调查中桡足类共检出14属,包含华哲水蚤属和中剑水蚤属。eDNA样品中共检出轮虫动物门17属,其中臂尾轮属、异尾轮属等12属在以往对广州水体的调查中也有发现[25-26],仅间盘轮属(Dissotrocha)、沼轮虫属(Limnias)、囊足轮属、猪吻轮属(Dicranophorus)和Lindia属未曾有报道。
利用eDNA技术对白洋淀无脊椎动物的调查中,共发现8门无脊椎动物,且以节肢动物门和轮虫动物门为主[27],与之相比,广州海珠国家湿地公园海珠湖无脊椎动物还检出了缓步动物门,组成更加丰富,可能是由于海珠湖的生境更加多样化。利用eDNA技术对太湖流域底栖动物调查检出底栖动物3门73属,包括环节动物门、软体动物门和节肢动物门[10],本次海珠湖检出无脊椎动物9门93属,以环节动物门、节肢动物门和轮虫动物门为主。
3.2 eDNA技术与传统形态鉴定的比较与形态学方法相比,eDNA技术对于某些群体分类的分辨率更高[28]。鸭绿江浮游动物群落结构调查中,发现eDNA与形态学鉴定方法在评价物种多样性方面具有较好的可比性,且eDNA技术在物种鉴定上具有明显优势[29]。本研究利用eDNA和形态学方法对海珠湖浮游动物种类进行鉴定和比较,共发现浮游动物43属。轮虫共发现24属,其中旋轮属、聚花轮属(Conochilus)、三肢轮属(Filinia)、镜轮属(Testudinella)、晶囊轮属(Asplanchna)、臂尾轮属、龟甲轮属、腔轮属(Lecane)、疣毛轮属和异尾轮属等10属为形态鉴定和eDNA鉴定的共有属;仅在形态学鉴定中发现和仅由eDNA技术所识别的浮游动物均只有7种,两者的相似性极高,具有很强的可比性。eDNA技术共发现桡足类14属,形态学仅鉴定出4属4种桡足类,eDNA技术在桡足类物种鉴定上具有更高的物种覆盖度和分辨能力,更能揭示“隐藏的”生物多样性[30]。
在海珠湖eDNA技术的研究中,大量外肛动物门、软体动物门、腹毛动物门等被检测出,填补了海珠湖部分微型生物调查的空白,表明了eDNA技术具有生物多样性监测的能力[31]。本次eDNA中检测出的囊足轮属、锚头鱼蚤属(Lernaea)等,在以往的海珠湖调查中从未见到,eDNA技术对传统形态学调查具有补充作用[8]。
eDNA技术未检出枝角类,仅在形态学鉴定中发现枝角类5属,这可能是因为条形码数据库不够完善,或引物18S rRNA基因V4区引物的特异性有关,需要结合其他的条形码弥补缺失的信息[32]。eDNA技术鉴定的结果中包含美国特有的Lampsili属、Adula属和分布在海洋的Placida属等,这些无脊椎动物的检出较为异常。出现异常结果可能与数据库错配,出现假阳性结果有关[20];本地物种的条形码数据库不够完善,数据库缺乏相应的物种序列[1, 33],从而没有相应的本地种序列匹配,导致结果异常;还可能是因为无脊椎动物的进化速度较快,致使条形码数据库的作用有限[34];另外,出现低盐海洋物种可能与潮汐有关,海珠湖位于珠江口,珠江口咸水上溯可能带来部分河口低盐种类[35-36]。数据库的不完整和准确率较低是使用eDNA技术的重大障碍,本地物种的条形码数据库构建极为重要[37]。因此,在现阶段采用形态学鉴定与eDNA相结合的方法能更加全面的揭示生物多样性,随着eDNA技术的进步和本土数据库的完善,eDNA技术会更加精准和可靠。
3.3 采样方式对eDNA技术结果的影响本次调查共采用3种方法采集eDNA样品。水采样品共检出50属68个OTUs,网采样品共检出27属35个OTUs,水采样品无脊椎动物丰富度高于网采样品;水采样品中发现节肢动物门15属,网采样品中仅4属;且外肛动物门、扁形动物门和缓步动物门仅在水采样品中检出,可能是因为其个体较小或丰度较低,导致网采过程中漏采[38]。不同浮游动物采样方法(直接过滤法和网富集法)对eDNA技术监测结果存在影响[39]。样品进行预过滤和不进行预过滤处理的eDNA样品所检测到的分类组成存在显著差异,进行预过滤的样品检测到的分类群更少[40]。
沉积物样品中检出70属103个OTUs,比水体样品(水采样品和网采样品)中无脊椎动物的丰富度更高。沉积物样品中发现线虫动物门27属,远高于水采样品和网采样品(分别检出5属和2属)。许多无脊椎动物种类将休眠作为一种生存策略,当环境较为恶劣时以休眠卵的形式存在和度过艰难时期[38, 41-42],并随着水流而分散,因此休眠卵库的多样性与无脊椎动物群落相似,甚至比水体更为丰富和多样[43]。当生境恶化,水体中的某些无脊椎动物便无法生存,将依靠休眠卵在适宜的条件下萌发重新建立种群,因此沉积物中休眠卵的丰富度可能会高于水体中的现存无脊椎动物[44]。除此之外,实验中使用蔗糖溶液对沉积物进行浮选,将浮选出的样品进行后续DNA提取等实验,在蔗糖溶液浮选过程中,节肢动物门(如桡足类、枝角类等)、轮虫动物门、外肛动物门、线虫动物门等无脊椎动物的卵容易从沉积物中分离出来[18],但浮选后的沉积物休眠卵样品不可避免的包含一些无脊椎动物残体、分泌物或粪便等,因此沉积物休眠卵样品中所检出的无脊椎动物并非全是休眠卵。
3.4 无脊椎动物群落结构对环境因子的响应海珠湖位于广州市区,经过几十年的快速发展,海珠湖及其周边水文环境均受到不同程度的影响[45-46],海珠湖也发展成为富营养化水体[47-48]。海珠湖无脊椎动物的优势属(臂尾轮属、龟甲轮属等,图 8),大多为小型浮游动物,群落结构有小型化的趋势。研究表明,水体富营养化可能导致浮游动物的小型化[49-50]。本次检出的异尾轮虫、疣毛轮虫、龟甲轮虫、裸腹溞等常见无脊椎动物,具有较强的生存能力和耐污能力[51],大多是富营养化水体的指示种类[52-53]。
无脊椎动物群落分布受所有环境因子的综合影响。调查发现,营养盐浓度是影响无脊椎动物分布的重要因子[54],如亚硝酸盐、总磷和磷酸盐等营养盐,通过影响浮游植物的生长来影响以浮游植物为食的无脊椎动物[55]。RDA分析表明,亚硝酸盐是影响网采无脊椎动物最重要的环境因子,新疆伊犁河谷上游水库的研究也发现亚硝酸盐与无脊椎动物呈正相关[56]。电导率是水体重要的理化指标之一,反映了水中离子的含量,其变化与水中营养盐的改变相关,电导率的大小反映了水中离子状态营养盐的多少,也是影响无脊椎动物群落结构的重要环境因子[57]。RDA分析表明,电导率是影响水采无脊椎动物最重要的环境因子,黄河三角洲和黄河口的无脊椎动物均与电导率呈正相关[58]。
4 附录附表Ⅰ~Ⅳ见电子版(DOI: 10.18307/2023.0437)。
| 附表Ⅰ 基于eDNA技术鉴定的海珠湖无脊椎动物名录 Appendix Ⅰ List of invertebrates at Haizhu Lake based on eDNA technique |
| 附表Ⅱ 环境因子与无脊椎动物门水平RDA分析结果——水采样品 Appendix Ⅱ Results of RDA analysis between invertebrates on phylum level and environmental factors - samples directly collected from water |
| 附表Ⅲ 环境因子与无脊椎动物门水平RDA分析结果——网采样品 Appendix Ⅲ Results of RDA analysis between invertebrates on phylum level and environmental factors - samples collected by a planktonic net |
| 附表Ⅳ 基于eDNA和形态学鉴定的浮游动物 Appendix Ⅳ Zooplankton identified based on eDNA technique and morphological identification |
致谢: 感谢林志斌、尹天齐、孙平宇、郭键林和梁俊策在样品采集方面给予的帮助,感谢汪文博在数据分析方面给予的帮助,感谢刘顺在文章写作方面给予的建议。
| [1] |
Wang M, Yuan Y, Yu HY et al. Construction of barcode library of freshwater macroinvertebrate in China. Environmental Monitoring in China, 2022, 38(1): 36-44. [王萌, 苑艺, 于海燕等. 中国淡水大型底栖无脊椎动物条形码数据库构建. 中国环境监测, 2022, 38(1): 36-44.] |
| [2] |
Vehkaoja M, Niemi M, Väänänen VM. Effects of urban infrastructure on aquatic invertebrate diversity. Urban Ecosystems, 2020, 23(4): 831-840. DOI:10.1007/s11252-020-00947-x |
| [3] |
Yang YF, Wang Q, Chen JF et al. Research advance in estuarine zooplankton ecology. Acta Ecologica Sinica, 2006, 26(2): 72-75. [杨宇峰, 王庆, 陈菊芳等. 河口浮游动物生态学研究进展. 生态学报, 2006, 26(2): 72-75. DOI:10.3321/j.issn:1000-0933.2006.02.034] |
| [4] |
de Necker L, Brendonck L, van Vuren J et al. Aquatic invertebrate community resilience and recovery in response to a Supra-Seasonal drought in an ecologically important naturally Saline Lake. Water, 2021, 13(7): 948. DOI:10.3390/w13070948 |
| [5] |
Wilhelm S, Knudsen. Species-specific detection and quantification of environmental DNA from marine fishes in the Baltic Sea. Journal of Experimental Marine Biology and Ecology, 2019, 510: 31-45. DOI:10.1016/j.jembe.2018.09.004 |
| [6] |
Qin CX, Zuo T, Yu G et al. Advances in research of environmental DNA (eDNA) in biomass assessment of aquatic ecosystems. South China Fisheries Science, 2020, 16(5): 123-128. [秦传新, 左涛, 于刚等. 环境DNA在水生生态系统生物量评估中的研究进展. 南方水产科学, 2020, 16(5): 123-128. DOI:10.12131/20190256] |
| [7] |
Reinholdt JM, Egelyng E, Agersnap S et al. Seasonal turnover in community composition of stream-associated macroinvertebrates inferred from freshwater environmental DNA metabarcoding. Environmental DNA, 2021, 3(4): 861-876. DOI:10.1002/edn3.193 |
| [8] |
BushA, Compson ZG, Monk WA et al. Studying ecosystems with DNA metabarcoding: Lessons from biomonitoring of aquatic macroinvertebrates. Frontiers in Ecology and Evolution, 2019, 7: 434. DOI:10.3389/fevo.2019.00434 |
| [9] |
Brantschen J, Blackman RC, Walser JC et al. Environmental DNA gives comparable results to morphology-based indices of macroinvertebrates in a large-scale ecological assessment. PLoS One, 2021, 16(9): e0257510. DOI:10.1371/journal.pone.0257510 |
| [10] |
Jin K, Zhang LJ, Zhang W et al. Monitoring and ecological health assessment of zoobenthos in Taihu Lake Basin based on environmental DNA macro-bar code. Environmental Monitoring in China, 2022, 38(1): 175-188. [金珂, 张丽娟, 张伟等. 基于环境DNA宏条形码的太湖流域底栖动物监测与生态健康评价. 中国环境监测, 2022, 38(1): 175-188.] |
| [11] |
Crane LC, Goldstein JS, Thomas DW et al. Effects of life stage on eDNA detection of the invasive European green crab (Carcinus maenas) in estuarine systems. Ecological Indicators, 2021, 124: 107412. DOI:10.1016/j.ecolind.2021.107412 |
| [12] |
Saccò M, Guzik MT, van der Heyde M et al. eDNA in subterranean ecosystems: Applications, technical aspects, and future prospects. Science of the Total Environment, 2022, 820: 153223. DOI:10.1016/j.scitotenv.2022.153223 |
| [13] |
Fernández S, Rodríguez S, Martínez JL et al. Evaluating freshwater macroinvertebrates from eDNA metabarcoding: A River Nalón case study. PLoS One, 2018, 13(8): e0201741. DOI:10.1371/journal.pone.0201741 |
| [14] |
Beentjes KK, Speksnijder AGCL, Schilthuizen M et al. Increased performance of DNA metabarcoding of macroinvertebrates by taxonomic sorting. PLoS One, 2019, 14(12): e0226527. DOI:10.1371/journal.pone.0226527 |
| [15] |
Zhu MM, Fan CX, Wu ZK et al. Temporal and spatial changes of phytoplankton community structure in Haizhu national wetland park. Chinese Journal of Ecology, 2020, 39(5): 1501-1508. [朱明明, 范存祥, 吴中奎等. 海珠国家湿地公园浮游植物群落结构时空变化. 生态学杂志, 2020, 39(5): 1501-1508. DOI:10.13292/j.1000-4890.202005.026] |
| [16] |
Huang YH, Fan CX, Wu ZK et al. Preliminary study on species composition and distribution of macrobenthos community in Haizhu wetland of Guangzhou. Wetland Science, 2020, 18(2): 200-206. [黄业辉, 范存祥, 吴中奎等. 广州市海珠湿地大型底栖动物群落物种组成和分布初探. 湿地科学, 2020, 18(2): 200-206.] |
| [17] |
国家环境保护总局《水和废水监测分析方法》编委会. 水和废水监测分析方法(第四版). 北京: 中国环境科学出版社, 2002.
|
| [18] |
Onbe T. Sugar flotation method for sorting the resting eggs of marine cladocerans and copepods from sea-bottom sediment. Nippon Suisan Gakkaishi, 1978, 44(12): 1411. DOI:10.2331/suisan.44.1411 |
| [19] |
Cheung MK, Au CH, Chu KH et al. Composition and genetic diversity of picoeukaryotes in subtropical coastal waters as revealed by 454 pyrosequencing. The ISME Journal, 2010, 4(8): 1053-1059. DOI:10.1038/ismej.2010.26 |
| [20] |
Li HX, Huang XN, Li SG et al. Environmental DNA (eDNA)-metabarcoding-based early monitoring and warning for invasive species in aquatic ecosystems. Biodiversity Science, 2019, 27(5): 491-504. [李晗溪, 黄雪娜, 李世国等. 基于环境DNA-宏条形码技术的水生生态系统入侵生物的早期监测与预警. 生物多样性, 2019, 27(5): 491-504. DOI:10.17520/biods.2018233] |
| [21] |
Coutant O, Richard-Hansen C, de Thoisy B et al. Amazonian mammal monitoring using aquatic environmental DNA. Molecular Ecology Resources, 2021, 21(6): 1875-1888. DOI:10.1111/1755-0998.13393 |
| [22] |
Euclide PT, Lor Y, Spear MJ et al. Environmental DNA metabarcoding as a tool for biodiversity assessment and monitoring: Reconstructing established fish communities of north-temperate lakes and rivers. Diversity and Distributions, 2021, 27(10): 1966-1980. DOI:10.1111/ddi.13253 |
| [23] |
Thomsen PF, Kielgast J, Iversen LL et al. Monitoring endangered freshwater biodiversity using environmental DNA. Molecular Ecology, 2012, 21(11): 2565-2573. DOI:10.1111/j.1365-294X.2011.05418.x |
| [24] |
Gao Y, Lai ZN, Zeng YY et al. Community structure of copepods and the relationship with aquatic environmental factors in the Pearl River Delta. Journal of Fishery Sciences of China, 2015, 22(2): 302-310. [高原, 赖子尼, 曾艳艺等. 珠江三角洲河网桡足类群落结构及其与水环境因子的关系. 中国水产科学, 2015, 22(2): 302-310. DOI:10.3724/SP.J.1118.2015.14211] |
| [25] |
Liang DW, Wang Q, Wei N et al. Spatial and temporal variation in rotifer community structure and the response to environmental factors among different water bodies in Guangzhou City. J Lake Sci, 2017, 29(6): 1433-1443. [梁迪文, 王庆, 魏南等. 广州市不同类型水体轮虫群落结构的时空变动及与理化因子间的关系. 湖泊科学, 2017, 29(6): 1433-1443. DOI:10.18307/2017.0615] |
| [26] |
Wang Q, Yang YF. Primary study on rotifer community structure in the Guangzhou segment of the river pearl. Acta Hydrobiologica Sinica, 2007, 31(2): 233-239. [王庆, 杨宇峰. 珠江广州河段轮虫群落结构的初步研究. 水生生物学报, 2007, 31(2): 233-239. DOI:10.3321/j.issn:1000-3207.2007.02.014] |
| [27] |
Wang XY, Wang Q, Yang YF et al. Comparison of invertebrate diversity in lake waters and their resting eggs in sediments, as revealed by high-throughput sequencing (HTS). Knowledge & Management of Aquatic Ecosystems, 2020(421): 19. DOI:10.1051/kmae/2020011 |
| [28] |
Banerji A, Bagley M, Elk M et al. Spatial and temporal dynamics of a freshwater eukaryotic plankton community revealed via 18S rRNA gene metabarcoding. Hydrobiologia, 2018, 818(1): 71-86. DOI:10.1007/s10750-018-3593-0 |
| [29] |
Gao YC, Li HT, Wang XC et al. Research on zooplankton diversity using DNA-based metabarcoding technique: A case study in the Yalvjiang Estuary. Acta Ecologica Sinica, 2020, 40(11): 3822-3832. [高养春, 李海涛, 王孝程等. 利用宏DNA条形码研究浮游动物多样性——以鸭绿江口为例. 生态学报, 2020, 40(11): 3822-3832. DOI:10.5846/stxb201903100451] |
| [30] |
Wang M, Jin XW, Lin XL et al. Advances in the macrozoobenthos biodiversity monitoring and ecosystem assessment using environmental DNA metabarcoding. Acta Ecologica Sinica, 2021, 41(18): 7440-7453. [王萌, 金小伟, 林晓龙等. 基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展. 生态学报, 2021, 41(18): 7440-7453. DOI:10.5846/stxb202009162411] |
| [31] |
Wang C, Tao M, Li AM et al. Research on the biodiversity of Qinhuai River based on environmental DNA metabacroding. Acta Ecologica Sinica, 2022, 42(2): 611-624. [王晨, 陶孟, 李爱民等. 基于环境DNA宏条形码技术的秦淮河生物多样性研究. 生态学报, 2022, 42(2): 611-624. DOI:10.5846/stxb202007221920] |
| [32] |
Duarte S, Leite B, Feio M et al. Integration of DNA-based approaches in aquatic ecological assessment using benthic macroinvertebrates. Water, 2021, 13(3): 331. DOI:10.3390/w13030331 |
| [33] |
Valentini A, Taberlet P, Miaud C et al. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [34] |
Yin TQ, Wang Q, Yang YF et al. Comparative study on zooplankton community structure in Pearl River Estuary based on morphological and DNA identification. Journal of Tropical Oceanography, 2022, 41(3): 172-185. [尹天齐, 王庆, 杨宇峰等. 基于形态学和DNA分子鉴定的珠江口浮游动物群落结构比较研究. 热带海洋学报, 2022, 41(3): 172-185. DOI:10.11978/2021151] |
| [35] |
Srichandan S, Baliarsingh SK, Lotliker AA et al. Unravelling tidal effect on zooplankton community structure in a tropical estuary. Environmental Monitoring and Assessment, 2021, 193(6): 1-21. DOI:10.1007/s10661-021-09112-z |
| [36] |
Huang BB, Zheng SX, Tian FG. Community dynamics of meso-and micro-zooplankton in the Pearl River Estuary in dry and wet seasons. Journal of Applied Oceanography, 2019, 38(1): 43-52. [黄彬彬, 郑淑娴, 田丰歌. 珠江口枯水期和丰水期中小型浮游动物群落动态. 应用海洋学学报, 2019, 38(1): 43-52. DOI:10.3969/J.ISSN.2095-4972.2019.01.005] |
| [37] |
Yang JH, Zhang XW, Zhang WW et al. Indigenous species barcode database improves the identification of zooplankton. PLoS One, 2017, 12(10): e0185697. DOI:10.1371/journal.pone.0185697 |
| [38] |
Chen HJ, Yu H, Liu GX. Comparison of copepod collection efficiencies by three commonly used plankton nets: A case study in Bohai Sea, China. Journal of Ocean University of China, 2016, 15(6): 1007-1013. DOI:10.1007/s11802-016-3122-6 |
| [39] |
Zhang JW, Yang JH, Zhang XW. Study on methods of zooplankton sampling for DNA metabarcoding monitoring. Environmental Monitoring and Forewarning, 2022, 14(1): 35-40. [张靖雯, 杨江华, 张效伟. 浮游动物DNA宏条形码多样性监测采样方法研究. 环境监控与预警, 2022, 14(1): 35-40.] |
| [40] |
Djurhuus A, Pitz K, Sawaya NA et al. Evaluation of marine zooplankton community structure through environmental DNA metabarcoding. Limnology and Oceanography: Methods, 2018, 16(4): 209-221. DOI:10.1002/lom3.10237 |
| [41] |
Radzikowski J. Resistance of dormant stages of planktonic invertebrates to adverse environmental conditions. Journal of Plankton Research, 2013, 35(4): 707-723. DOI:10.1093/plankt/fbt032 |
| [42] |
Wang Q, Luan LL, Chen LD et al. Advances in copepod resting egg ecology in estuarine and coastal waters. Chinese Journal of Applied Ecology, 2015, 26(7): 2213-2224. [王庆, 栾磊磊, 陈亮东等. 河口近海桡足类休眠卵生态学研究进展. 应用生态学报, 2015, 26(7): 2213-2224. DOI:10.3321/j.issn:1001-0742.2007.06.017] |
| [43] |
Chaparro G, Mariani M, Hein T. Diversity of dormant and active zooplankton stages: Spatial patterns across scales in temperate riverine floodplains. Journal of Plankton Research, 2021, 43(1): 61-71. DOI:10.1093/plankt/fbaa063 |
| [44] |
Patterson LN, Harris BD, Covi JA. Lack of dormancy to protect diversity: Decrease in diversity of active zooplankton community observed in lake with depauperate egg bank. Science of the Total Environment, 2020, 723: 138074. DOI:10.1016/j.scitotenv.2020.138074 |
| [45] |
Cheng YH, Mao YP, Zhang H. Characteristics of anthropogenic net input of nitrogen and phosphorus and suggestions on pollution control in Pearl River Delta. Chinese Journal of Environmental Engineering, 2022, 16(6): 2049-2060. [程元辉, 毛宇鹏, 张洪. 珠江三角洲地区人为氮磷净输入特征及污染管控建议. 环境工程学报, 2022, 16(6): 2049-2060. DOI:10.12030/j.cjee.202111035] |
| [46] |
Zeng DN, Niu LX, Tao W et al. Nutrient dynamics in Pearl River Estuary and their eutrophication evaluation. Journal of Guangdong Ocean University, 2020, 40(3): 221-227. [曾丹娜, 牛丽霞, 陶伟等. 夏季珠江口水域营养盐分布特征及其富营养化评价. 广东海洋大学学报, 2020, 40(3): 221-227.] |
| [47] |
Tang MY, Zhou W, Qiao YM et al. Forms of phosphorus in sediments of major lakes in Guangzhou. Journal of Hydroecology, 2023, 44(2): 54-63. [唐梦瑶, 周雯, 乔永民等. 广州市主要湖泊沉积物磷的赋存形态. 水生态学杂志, 2023, 44(2): 54-63. DOI:10.15928/j.1674-3075.202111300399] |
| [48] |
Xiu C, Xie L, Xiao RB. Spatial-temporal variations of water quality parameters in Haizhu wetland in Guangzhou and the water protection strategy. Guangdong Chemical Industry, 2018, 45(15): 176-178. [修晨, 谢琳, 肖荣波. 广州海珠湿地2015—2016年水质时空变化特征及保护策略. 广东化工, 2018, 45(15): 176-178.] |
| [49] |
Chen X, Li Z, Tang FJ et al. Zooplankton community structure and its relationship with environmental factors in Lianhuan Lake. Journal of Hydroecology, 2020, 41(6): 89-97. [陈昕, 李喆, 唐富江等. 连环湖两水体浮游动物群落结构特征及水质评价. 水生态学杂志, 2020, 41(6): 89-97. DOI:10.15928/j.1674-3075.2020.06.011] |
| [50] |
Lan B, He LP, Huang YJ et al. Tempo-spatial variations of zooplankton communities in relation to environmental factors and the ecological implications: A case study in the hinterland of the Three Gorges Reservoir area, China. PLoS One, 2021, 16(8): e0256313. DOI:10.1371/journal.pone.0256313 |
| [51] |
Onwudinjo CC, Egborge ABM. Rotifers of Benin River, Nigeria. Hydrobiologia, 1994, 272(1/2/3): 87-94. DOI:10.1007/BF00006514 |
| [52] |
日本生态学会环境问题专门委员会. 环境和指示生物. 北京: 中国环境科学出版社, 1987.
|
| [53] |
Chen GR, Zhong P, Zhang XF et al. Zooplankton and its relationship with water quality in Huizhou West Lake. J Lake Sci, 2008, 20(3): 351-356. [陈光荣, 钟萍, 张修峰等. 惠州西湖浮游动物及其与水质的关系. 湖泊科学, 2008, 20(3): 351-356. DOI:10.18307/2008.0314] |
| [54] |
Li Y, Wang R, Su HJ et al. Eutrophication and predation mediate zooplankton diversity and network structure. Limnology and Oceanography, 2021, 67: S133-S145. DOI:10.1002/lno.11957 |
| [55] |
Glibert PM, Al-Azri A, Icarus Allen J et al. Key questions and recent research advances on harmful algal blooms in relation to nutrients and eutrophication. Ecological Studies. Cham: Springer International Publishing, 2018: 229-259. DOI: 10.1007/978-3-319-70069-4.
|
| [56] |
Peng L, Ru HJ, Tao L et al. Zooplankton distribution and water environment characteristics in reservoir of upper Ili River Valley: A case study in Qapchy Lake. Arid Land Geography, 2022, 45(2): 535-545. [彭亮, 茹辉军, 陶玲等. 伊犁河上游河谷水库浮游动物分布及水环境特征——以恰甫其海为例. 干旱区地理, 2022, 45(2): 535-545.] |
| [57] |
Liu DD, Wu HT, Lu KL et al. Effects of spatial and environmental factors on benthic invertebrate communities in natural and freshwater restored wetlands of the Yellow River Delta. Acta Ecologica Sinica, 2021, 41(17): 6893-6903. [刘丹丹, 武海涛, 芦康乐等. 空间和环境因子对黄河口自然和淡水恢复湿地底栖动物群落的差异影响. 生态学报, 2021, 41(17): 6893-6903. DOI:10.5846/stxb202009052312] |
| [58] |
Lu KL, Yang MY, Wu HT et al. Composition of benthic invertebrate communities in reed (Phragmites australis) wetlands of the Yellow River Delta: Comparisons between oil exploitation and freshwater recharge areas. Acta Ecologica Sinica, 2020, 40(5): 1637-1649. [芦康乐, 杨萌尧, 武海涛等. 黄河三角洲芦苇湿地底栖无脊椎动物与环境因子的关系研究——以石油开采区与淡水补给区为例. 生态学报, 2020, 40(5): 1637-1649. DOI:10.5846/stxb201901100087] |
 2023, Vol. 35
2023, Vol. 35