摘要
近年来,水利水电工程中暴发了沼蛤(淡水壳菜)污损问题,不同程度地影响了水利和水电工程的输水、生产等安全,引起了国内外社会广泛关注。本文研究结果显示,沼蛤在长江流域主要分布于四川及以下的低海拔区,四川以上高海拔区未见记录;基于COI和ITS1基因联合构建的系统发育树分析显示,长江流域沼蛤可能存在两个类群,其中COI序列A+T含量(64.7%)显著高于C+G含量(35.3%),种内遗传距离范围为0~0.123;COI序列总单倍型多样性指数为0.893,核苷酸多样性指数为0.069;ITS1序列A+T含量为45.3%,C+G含量为54.7%,种内遗传距离范围为0~0.061;ITS1序列总单倍型多样性指数为0.679,核苷酸多样性指数为0.028。基于COI基因分析的两个亲缘关系类群间的遗传分化系数为0.844,基因差异分化系数为0.118;基于ITS1基因分析的相应参数分别为0.927和0.492。基于形态学特征分析显示,长江流域沼蛤的长高比、长宽比和韧带长度差异显著,可明显分成两个类群,且与COI和ITS1基因分析显示的两个类群相对应。基于栖息生境分析显示,长江流域沼蛤类群1主要生活在水流较缓、底质以卵石为主的区域,表现为缓流型;类群2主要生活在水流较急、底质以泥沙为主的区域,表现为激流型,对环境的适应性更强,分布范围较类群1更广,在传播扩散能力方面表现更强。本研究有助于了解长江流域沼蛤基础生物学信息,可为流域沼蛤的综合防控提供重要参考。
Abstract
In recent years, the problem of biological fouling by Limnoperna fortune (Dunker, 1857) has occurred in water resources and hydraulic engineering, affecting the safety of water transmission and production of water conservancy and hydropower projects to varying degrees. The problem has attracted extensive attention from the domestic and foreign communities. This study showed that L. fortunei in the Yangtze River Basin was mainly distributed in the low altitude area of Sichuan and below. No record was found in the high-altitude area above Sichuan. Phylogenetic tree analysis utilizing COI and ITS1 genes revealed two groups of L. fortunei in the Yangtze River Basin. The A+T content of COI sequences was notably higher at 64.7% compared to the C+G content at 35.3%, with intraspecific genetic distances ranging from 0 to 0.123. The total haplotype diversity index for COI sequences was 0.893. The nucleotide diversity index was 0.069. For the ITS1 sequences, the A+T content was 45.3% and the C+G content was 54.7%, with intraspecific genetic distances ranging from 0 to 0.061. The total haplotype diversity index for ITS1 sequences was 0.679. The nucleotide diversity index was 0.028. Genetic differentiation analysis based on the COI gene yielded a coefficient of genetic differentiation of 0.844 and a coefficient of differential gene differentiation of 0.118 between the two related groups. Similarly, analysis based on the ITS1 gene resulted in a coefficient of genetic differentiation of 0.927 and a coefficient of differential gene differentiation of 0.492. The morphological characteristics and habitat analysis showed that there were significant differences in the morphological characteristics and habitats of the two ecotypes of L. fortunei in the Yangtze River basin, which can be clearly divided into two groups, i.e., Group 1 and Group 2 as shown in COI and ITS1 gene analysis. The length-height ratio, length-width ratio and ligament length of the two groups were significantly different. Group 1 mainly lived in the areas with slow flow and pebble-based bottom, showing a slow flow type. Group 2 mainly lived in the areas with fast flow and sand-based bottom, showing a rapid flow type, and had stronger adaptability to the environment. It had a wider distribution range than Group 1. This study improved our understanding of the basic biological information of L. fortunei in the Yangtze River basin, and provided a reference for the comprehensive prevention and control of L. fortunei in the basin.
Keywords
沼蛤Limnoperna fortunei(Dunker,1857),俗称淡水壳菜,隶属于软体动物门双壳纲贻贝目贻贝科沼蛤属[1-3],是淡水生态系统中大型底栖动物重要类群之一,广泛分布于自然河湖水体,在水生态系统中具有重要的生态功能[4-7]。沼蛤具有生命周期短、成长快、繁殖强的特点,幼体营浮游生活,能够随着水流快速扩散,是其适应扩散的重要原因[8-9];成体营固着生活,通过足丝这一特殊的结构实现附着,特别难以清除,是造成水利水电工程中水工构筑物生物污损的重要原因[10-11]。国内外有关沼蛤生物污损问题的报道较多,尤其是大型调水工程、水力发电站等[12-14]。相关研究显示温度、光照、流速、pH值、附着基质等是影响沼蛤生长繁殖及分布的关键因素[3,15-16]。基于此构建了生物、物理、化学等技术原理的综合防控措施,效果不一[9-10,17]。截止目前,沼蛤的污损防治仍旧是世界性难题,尤其是在热带及亚热带区域污损防治难度更大。
国内外沼蛤的分类均主要依据其外壳形态特征,《中国经济动物志》[18]中记载沼蛤属在国内仅有一种,即沼蛤;马佳卉等[19]提到中国目前发现的贻贝目种类有73种,其中仅l种为淡水分布,即沼蛤。贻贝的栖息地环境条件差异很大,贻贝为了适应这些不同的环境而不断分化,这种适应性分化使得贻贝分布范围广泛,且种类繁多[20]。贻贝目中,与沼蛤属近似属种均存在于海洋中,中国记录的有30多种,主要有贻贝属的紫贻贝Mytilus galloprovincialis(Lamarck,1819)、翡翠贻贝Perna viridis(Linnaeus,1758)以及厚壳贻贝Mytilus coruscus(Gould,1861)等。沼蛤与欧洲入侵生物斑马贻贝Dreissena polymorpha(Pallas,1771)有较多相似特征,例如壳长大小相近、生长迅速、依靠足丝进行附着、雌雄异体和体外受精进行繁殖等[10]。贻贝科的物种因为外部形态相似等,使得科内物种的分类、分布及扩散研究面临挑战[21]。随着分子生物学技术的发展,DNA条形码等新技术逐渐应用到贻贝的种类鉴定中,也证实紫贻贝和贻贝在遗传上存在显著差异[22-24]。长江流域面积约180万km2,横跨中东西部,水系发达,沼蛤在流域也广泛分布,但关于其多样性的研究仍未开展,为其防控可提供的基础生物学资料缺乏。
基于此,本研究通过对长江流域及南水北调中线范围地区的沼蛤标本进行采集,运用基因克隆和sanger测序技术测定各样本基因序列,结合国际数据库中沼蛤及其他贻贝属生物的基因序列,运用多种基因构建沼蛤系统发育树。在此基础上,结合标本的关键形态学数据和栖息生境特征,揭示不同环境中沼蛤的基因及形态多样性,为其在不同区域的综合防控提供基础生物学数据支撑。本文沼蛤样本记录区域包括:长江干流涉及重庆、湖北和安徽等江段,重要支流涉及汉江、湘江、沅江,通江湖泊涉及鄱阳湖,以及南水北调中线干渠。
1 材料与方法
1.1 标本采集和数据来源
本研究于2022年在长江流域以及南水北调中线干渠开展沼蛤样本采集,监测点位布设合计300个(图1A),其中60个点位采集到沼蛤样本(图1B)。采样严格按照底栖动物监测技术规范要求,即用1/16 m2采泥器采集4次,再加上边长0.3 m的三角拖网上下游各拖行5 m。在获取的标本中再选取有完整组织的样本,共计48个个体,其中干流5个、支流39个、通江湖泊1个、中线干渠3个,用75%酒精低温保存(附表Ⅰ),用于实验研究分析,并在NCBI和BOLD数据库下载已有沼蛤序列信息(附表Ⅱ),用于后续分析。
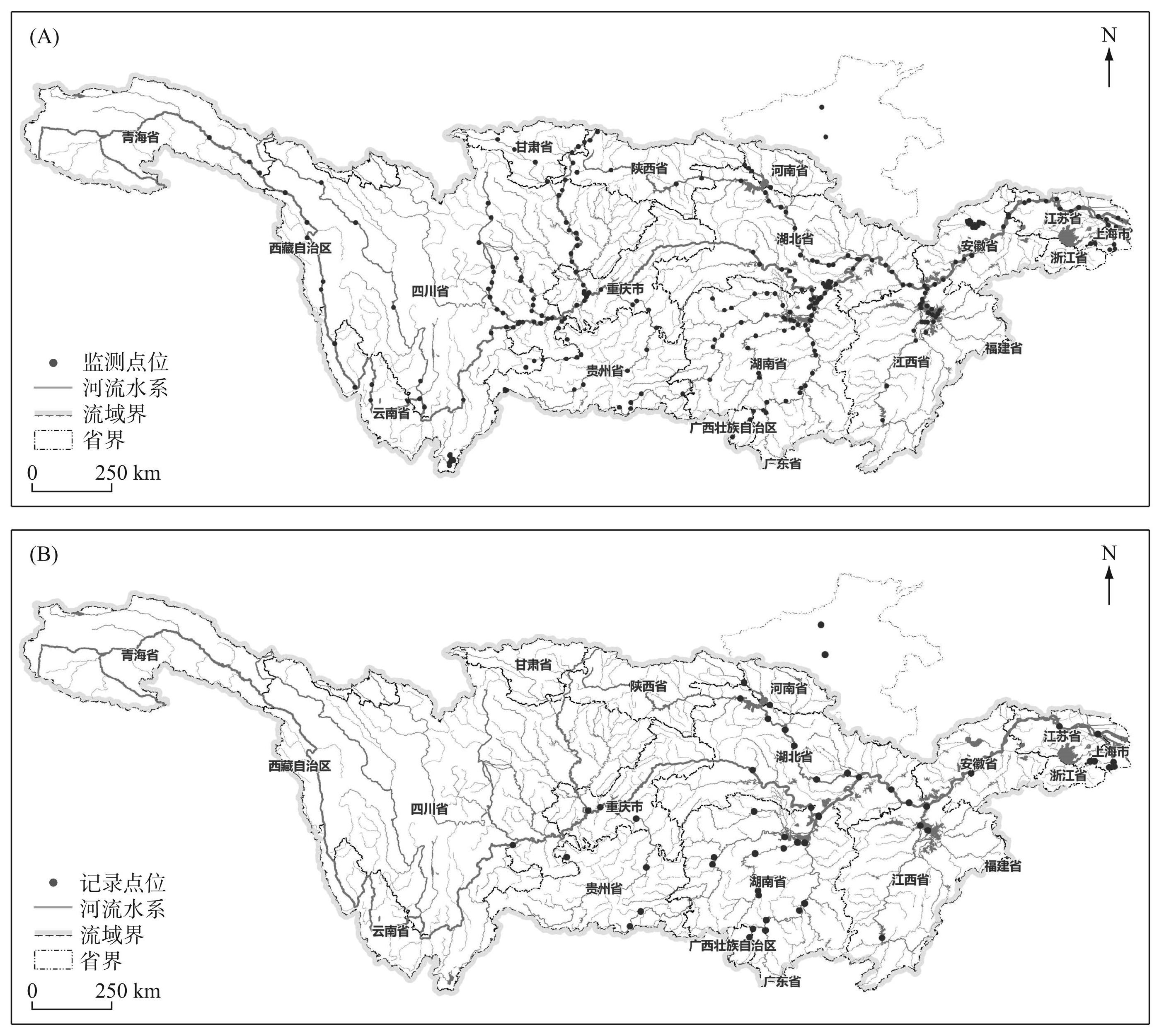
图1沼蛤监测点位(A)及记录点位(B) (该图基于自然资源部标准地图服务网站下载的审图号为GS(2023)2767号的标准地图制作,底图无修改)
Fig.1Monitoring sites (A) and recording sites (B) of L. fortunei
1.2 DNA提取和检测
采用酒精固定沼蛤肌肉组织,使用生工生物工程有限公司的DNA提取试剂盒进行DNA提取。PCR反应体系为30 μL:ddH2O 13 μL、模板DNA 1 μL、2× Taq Maste Mix 15 μL、上下游引物(COI和ITS1通用引物)各0.5 μL。COI上下游引物序列[25]:LCO1490:5′-GGTCAACAAATCATAAAGATATTGG-3′,HCO2198:5′-TAAACTTCAGGGTGACCAAAAAATCA-3′;ITS1上下游引物序列[26]:ITS1F:5′-TAACAAGGTTTCCGTAGGTG-3′,ITS4:5′-TCCTCCGCTTATTGATATGC-3′。PCR程序设置为:95℃预变性3 min、95℃变性30 s、56℃退火30 s、72℃延伸30 s,共35个循环,72℃延伸5 min。PCR扩增产物经1%琼脂糖凝胶电泳初步检测DNA片段的长度和完整度,使用美国ProteinSimple公司生产的“化学发光成像系统”凝胶电泳检测仪器进行成像,选择条带清晰明亮、无杂带、无拖尾的产物外送生物公司进行产物纯化及测序。
1.3 数据处理和分析
沼蛤的测序结果使用Seq Man软件[27]、Clustal X 1.83软件[28]和Bio Edit 7.0软件[29]进行序列的人工核查、组装拼接和初步比对,获得一致序列备用,最后提交至BLAST进行同源性比对分析。使用DAMBE 5.3软件进行碱基替换饱和度分析。
使用MEGA 7.0[30]软件计算序列中碱基组成和遗传距离等参数,基于邻接法(neighbor-joining,NJ),以物种Perna perna、Perna viridis和Perna indica作为外群,采用Kimura双参数模型,设置bootstrap值重复500次检验置信度,构建沼蛤COI和ITS1序列的NJ系统发育树,同时将两个基因串联进行联合分析建树。通过Dna SP 6.1软件[31]分析序列单倍型数(H)、核苷酸组成等。
1.4 形态和生境分析
沼蛤形态分析测量使用ZEISS体视显微镜,观察其壳体颜色,测量壳长、壳宽、壳高及韧带长度的相关数据(图2),进行长和高、长和宽的比值比较,使用Excel 2021绘制形态差异分析图,并检验显著性。沼蛤长、宽、高测量方法参照狭长贝类测量方法[32]。基于现场生境状况记录,比较分析不同样本的分布特点。

图2沼蛤形态测量指标 (A:韧带长度;B:壳高;C:壳长;D:壳宽)
Fig.2Morphometry indicators of L. fortunei (A: ligament length; B: shell height; C: shell length; D: shell width)
2 结果
2.1 序列特征和碱基替换分析
沼蛤COI和ITS1测序数据经组装拼接、比对和BLAST验证序列同源性后,共获得48条序列用于后续分析。经比对分析,沼蛤COI序列T、C、A、G碱基的平均含量分别为41.8%、12.8%、22.9%、22.5%,其中C+G含量为35.3%,A+T含量为64.7%,碱基T含量最高,C含量最低,核苷酸的组成明显偏离C,具有明显的AT碱基偏倚性。沼蛤ITS1序列T、C、A、G碱基的平均含量分别为25.2%、27.0%、20.1%、27.7%,其中C+G含量为54.7%,A+T含量为45.3%。DAMBE软件分析结果如表1可知,Iss<Iss.c,P<0.05,沼蛤COI和ITS1基因序列未达到饱和,可进行下一步系统发育的分析研究。
表1沼蛤COI和ITS1序列碱基替换饱和性分析
Tab.1 Analysis of base substitution saturation of COI and ITS1 sequences of L. fortunei

2.2 遗传距离和系统发育分析
基于COI和ITS1单基因的系统发育树分析结果,沼蛤可能存在遗传分化(附图Ⅰ、Ⅱ)。基于COI和ITS1序列遗传距离计算分析显示,COI遗传距离范围为0~0.123,ITS1遗传距离范围为0~0.061。基于COI、ITS1联合基因构建的沼蛤系统发育树,结果表明本研究沼蛤基因型存在显著差异(图3),这为沼蛤单系进化提供了理论支持。系统发育分析显示,沅江(YJHH-01、YJHH-02、YJX-02)、汉江(HJSY-01、HJHZ-01、HJHZ-02、HJJM-02、HJJM-03、HJXY-01、HJXY-02、HJXY-03、HJXY-04、HJWH-01、HJWH-02)、长江干流(CJCQ-01、CJTL-01)、资江(ZJYYX-01、ZJYY-01、ZJYY-02)、丹江(DJNY-01、DJNY-02)、湘江(XJHY-03)、澧水(LSCD-01)的个体COI和ITS1序列相似度高,亲缘关系较近,将其标记为长江流域类群1(Group 1);中线干渠(ZXGQNY-02、ZXGQPDS-01、ZXGQZZ-01)、湘江(XJXYGL-01、XJHY-02、XJHY-04、XJHY-05)、资江(ZJLD-01、ZJLD-02、ZJSY-01、ZJSY-02)、沅江(YJX-01)、汉江(HJJM-01、HJTM-01、HJTM-02)、赤水河(CSHZY-01)、长江干流(CJHS-01、CJTL-02、CJTL-03)、鄱阳湖(PYHNC-01)、汇中河(HZHSH-01、HZHSH-02)、中运河(ZYHSH-01、ZYHSH-02)、太浦河(TPHJX-01)个体序列相似度高,亲缘关系较近,标记为长江流域类群2(Group 2),且该类群的COI序列与已知的日本、韩国等地的序列相似度高;对南水北调中线干渠由南至北3个点位的沼蛤样品COI和ITS1序列分析发现,相似度在97%以上。

图3基于COI和ITS1联合基因构建的沼蛤NJ系统发育树
Fig.3Joint NJ phylogenetic tree based on COI and ITS1 sequences of L. fortunei
表2基于COI序列的沼蛤遗传信息参数
Tab.2 Genetic diversity parameters of COI sequences of L. fortunei

表3基于ITS1序列的沼蛤遗传信息参数
Tab.3 Genetic diversity parameters of ITS1 sequences of L. fortunei
2.3 遗传信息分析
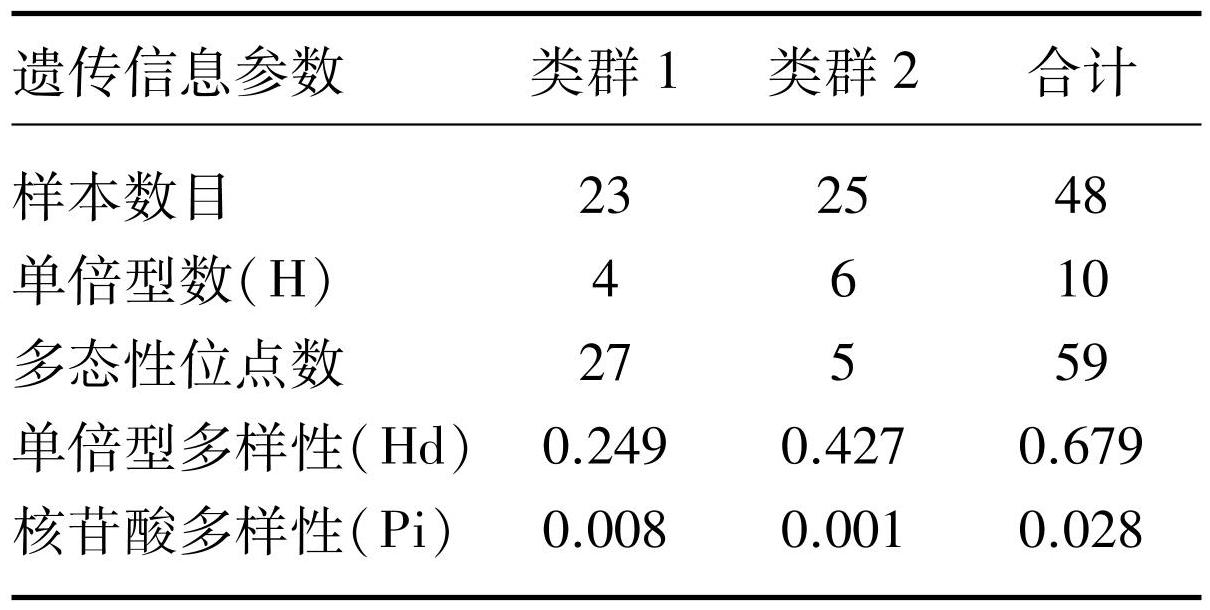
基于COI序列的遗传信息分析结果如表2所示,长江流域沼蛤存在18个单倍型,单倍型多样性(Hd)为0.893,核苷酸多样性指数(Pi)为0.069,检测到123个多态性位点,其中包含11个单一突变位点和112个简约信息位点。基于ITS1序列的遗传信息分析结果如表3所示,长江流域沼蛤存在10个单倍型, Hd为0.679,Pi为0.028,检测到59个多态性位点,其中包含4个单一突变位点和55个简约信息位点。本研究沼蛤2个亲缘类群间COI序列的遗传分化系数(Fst)为0.844,类群间的基因差异分化系数(Gst)为0.118,基因流(Nm)为0.093(P<0.05)。沼蛤2个亲缘类群间ITS1序列的Fst值为0.927,类群间的Gst值为0.492,Nm为0.040(P<0.05)。
2.4 形态和生境分析
基于系统发育分析分成的两类群所对应形态学比较观察分析显示,颜色差异不显著(图4),均表现出黄色、紫色及紫褐色,不能作为有效分类特征;沼蛤的贝壳长高比、长宽比和韧带长度存在显著差异(依次为P<0.001、P<0.05、P<0.001),可以明显分为两个类群(图5)。
基于本研究沼蛤所对应生境进一步分析发现,类群1生活在水流较缓、底质以卵石为主的区域,表现为缓流型;类群2生活在水流较急、底质以泥沙为主的区域,表现为激流型,分布范围较类群1更广,在传播扩散能力方面表现更强(表4)。

图4沼蛤贝壳颜色 (A和C:类群1颜色(黄色、紫色及紫褐色); B和D:类群2颜色(黄色、紫色及紫褐色))
Fig.4Shell colors of L. fortunei (A and C: Group 1 colors (yellow, purple and purple brown) ; B and D: Group 2 colors (yellow, purple and purple brown) )
3 讨论
Zhan等[33]通过COI聚类分析揭示了南美入侵沼蛤存在两个遗传上截然不同的聚类,这两个集群在南美的入侵范围显示出遗传上的不连续分布,从同一水系取样的种群没有被分配到一个单一的集群,而地理上孤立的群体往往被分配到相同的集群。三维因子对应分析证实了这一结果,将所有种群分为两类,同时基于微卫星的金贻贝种群分析支持南美的两个主要遗传群。他们推测沼蛤在入侵初期,两个集群的形成有3个可能的过程:1)两个集群是由两种不同的遗传来源入侵导致的;2)本地适应性强,进化迅速;3)群体变化引起遗传漂变。COI基因作为普遍应用的分子标记之一,庞宇杰等[34]比较了紫贻贝野生群体与养殖群体线粒体COI基因的遗传多样性,发现野生型个体多与养殖型交错聚在一起,说明乳山野生和养殖紫贻贝间基本无分化,亲缘关系较近。有报道表明,ITS1序列最短仅有70 bp[35],最长的有760 bp[36],所以许多学者把ITS1基因当作物种鉴定的有效分子标记[37]。目前基于ITS1基因在沼蛤系统进化方面的研究尚未报道,仅对其他软体动物进行了相关研究,如廖德杰等[38]利用核糖体ITS1序列及5S rDNA序列对4种扇贝进行了序列特征、遗传多样性、遗传距离及系统发育等分析研究,发现岩扇贝与虾夷扇贝的遗传距离最小,而岩扇贝与海湾扇贝遗传距离最大,且系统进化树分析显示岩扇贝与虾夷扇贝独自聚为一支。本研究基于所获得的COI和ITS1序列以及GenBank数据库中其他相应的序列,初步构建了长江流域沼蛤系统发育树,探讨了沼蛤的亲缘关系。基于COI和ITS1分子标记的系统发育树显示,长江流域沼蛤有2个亲缘关系类群,且分支间遗传距离较大(COI遗传距离范围为0~0.123,ITS1遗传距离范围为0~0.061),置信度较高。南水北调中线总干渠的沼蛤与汉江、长江干流、湘江等水体的部分沼蛤个体序列相似度高、亲缘关系接近。基于中线总干渠沼蛤的COI和ITS1基因分析,得出相似度在97%以上,显示干渠内沼蛤多样性低、种群单一。

图5沼蛤形态差异比较分析
Fig.5Morphological differences analysis of L. fortunei
表4本研究记录的沼蛤栖息生境
Tab.4 The habitats of L. fortunei recorded in this study
Grant等[39]将核苷酸多样性与单倍型多样性间的组合分为以下类型:1)较低的核苷酸多样性(Pi<0.005)和较低的单倍型多样性(Hd<0.5);2)较高的单倍型多样性和较高的核苷酸多样性;3)较高的单倍型多样性和较低的核苷酸多样性;4)较低的单倍型多样性和较高的核苷酸多样性。本研究遗传分析显示沼蛤的序列结果属于第2种类型。本研究中沼蛤的COI序列存在18个单倍型,Hd为0.893,Pi为0.069,检测到123个多态性位点,其中包含11个单一突变位点和112个简约信息位点;ITS1序列存在10个单倍型,Hd为0.679,Pi为0.028,检测到59个多态性位点,其中包含4个单一突变位点和55个简约信息位点。通常而言,水生生物群体中丰富的遗传信息可以提高应对栖息地变化和其他选择力的进化潜力[40]。综合本研究中COI和ITS1基因的分析结果,长江流域沼蛤对环境变化可能表现出较强的适应性及进化潜力。
沼蛤可以随着跨流域调水工程或商船进入新的环境[41],并迅速扩散。长江是中国第一大河流,地区性商业航运频繁,本研究记录的两个类群可能随着水流或附着于商船底部进入新的环境,因此存在同一点位出现两个类群的可能(图6)。因基因流不显著(COI为0.093,ITS1为0.040),来自不同的遗传分化可以保持下去,这一结果与Zhan等[33]报道的一致。此外,Zhan等[33]还报道了船媒“跳跃”传播是南美沼蛤传播的主要方式,而下游的自然传播对遗传模式的影响有限。不同的温度可能导致软体动物的产卵季节和存活率不同,从而导致这两个区域之间的基因流动不频繁[42]。长江流域纬度跨度范围比较大,各区域水温又有所差异,这可能是沼蛤基因流不显著的原因之一。事实上,在拉普拉塔河的不同沼蛤种群中发现了不同的产卵季节[43],长江流域沼蛤很可能也存在类似情况。从本研究沼蛤记录点位分布分析可知(图6),其主要集中分布于四川以下的长江流域中下游,整体海拔较低,且存在两个类群(类群1和类群2);在高海拔区域未发现沼蛤记录。类群1主要分布在长江中游及北部地区;类群2的分布范围较类群1更广,在长江以北和以南地区均有分布,入海口附近也有记录,类群2表现出对环境更强的适应性。系统发育分析也显示类群2与国外报道的沼蛤具有很高的相似性,说明适应性更强的类群2在传播扩散上能力可能更强。因此,后期沼蛤的防控则需重点关注适应性和传播能力表现更强的类群。
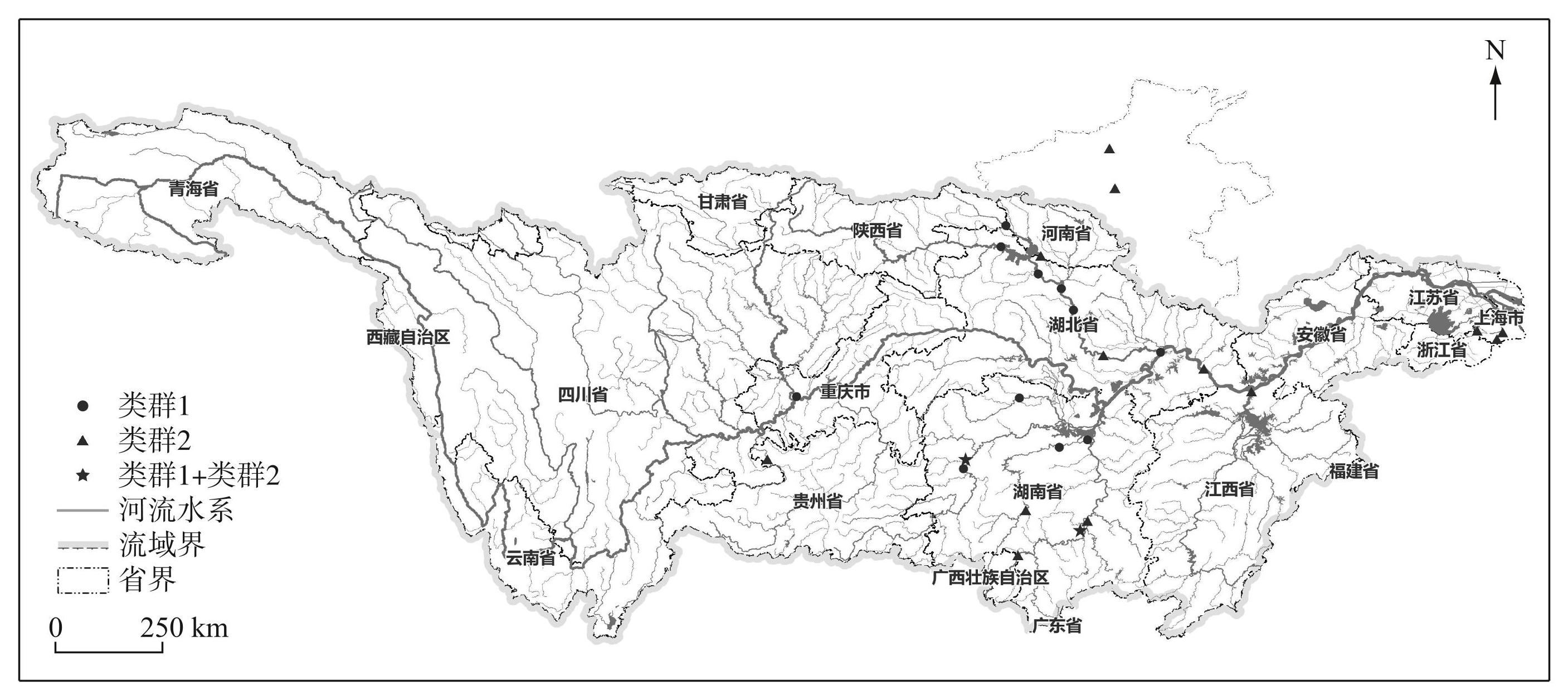
图6沼蛤两个类群分布 (该图基于自然资源部标准地图服务网站下载的审图号为GS(2023)2767号的标准地图制作,底图无修改)
Fig.6Distribution of the two goups of L. fortunei
遗传学研究认为,Fst值可用来估计亚种群间遗传分化,即用来衡量种群内亚种群之间近交程度[44]。根据Wright[45]的遗传分化系数划分标准,可分为如下4类:Fst值在0~0.05间,为无分化;Fst值在0.05~0.15间,为中度分化;Fst值在0.15~0.25间,为高度分化;Fst值>0.25,为重度分化。本研究长江流域沼蛤两个类群间COI序列的Fst值为0.844,Gst值为0.118,表现为重度遗传分化;ITS1序列的Fst值为0.927,Gst值为0.492,也表现为重度遗传分化,进一步证实了沼蛤群体之间可能存在遗传分化。Zhan等[33]利用COI基因和8个微卫星标记对南美22个入侵种群(N=712)进行了遗传分析,观察到较高的遗传分化。de Souza等[46]利用自行开发的13个微卫星标记对来自3个水库群体的48个沼蛤个体进行遗传多样性和种群遗传结构分析,结果表明各组间的Fst值较低且不显著(P>0.05),可能在引入新栖息地过程中导致遗传结构缺失。
综合长江干流及其主要支流、中线干渠记录的沼蛤形态分析显示,外壳颜色呈现不规律分布,不能作为有效区分的特征;两个类群的长高比、长宽比和韧带长度差异显著,可明显分为两个类群,且类群2的数值比类群1高,差异显著。基于沼蛤栖息生境分析显示,类群1有生活在水流较缓、底质以卵石为主的区域倾向,表现为缓流型;类群2有生活在水流较急、底质以泥沙为主的区域倾向,表现为激流型。形态和生境特征的分析结果与系统发育树结果保持一致,可明显分为两个类群。研究显示沼蛤对环境条件(如温度、盐度等)大幅度变化的适应性及其对外部威胁力量(如捕食、水流冲刷等)的适应能力强[41,47-48],成为沼蛤产生污损的基础,也给沼蛤污损防控带来挑战。本研究中类群2的激流型分布范围广泛,记录水系更多,能更好地适应流水环境,在全球贸易频繁的航运过程中可能更易广泛传播,进而引发全球沼蛤(淡水壳菜)生物污损问题。
4 结论
基于本研究分析可知,沼蛤(淡水壳菜)主要分布在四川及以下的低海拔区,四川以上高海拔区未见记录;南水北调中线总干渠的沼蛤与汉江、长江干流、湘江等水体的部分沼蛤序列相似度高、亲缘关系接近。基于沼蛤COI及ITS1的基因序列分析可知,长江流域沼蛤不是单一种,可分为两个类群(类群1和类群2)。基于已记录的沼蛤形态特征、栖息生境分析显示,长江流域沼蛤有偏向底质为泥沙生活及偏向于底质为卵石生活两种类群,其中类群1表现为缓流型,类群2表现为激流型。沼蛤激流型分布范围相对更广,对环境的适应性更强,在传播扩散上表现能力可能更强。后期沼蛤防控需重点关注适应性和传播能力强的类群2;后续研究会扩大调查区域,提高样本采集量,增加其他遗传标记,进一步加强沼蛤生活史、适应性机制等方面研究,以期进一步提升对该类群遗传多样性和扩散的认识,为其综合防控提供基础生物学信息支撑。
5 附录
附表Ⅰ、Ⅱ和附图Ⅰ、Ⅱ见电子版(DOI: 10.18307/2025.0133)。
